Model info
| Transcription factor | ETS1 (GeneCards) | ||||||||
| Model | ETS1_HUMAN.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 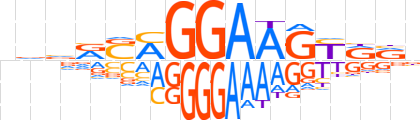 | ||||||||
| LOGO (reverse complement) | 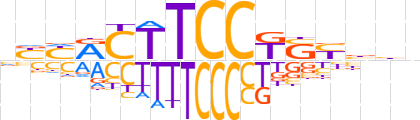 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 15 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | vvvRSMGGAAGYRRn | ||||||||
| Best auROC (human) | 0.966 | ||||||||
| Best auROC (mouse) | 0.94 | ||||||||
| Peak sets in benchmark (human) | 45 | ||||||||
| Peak sets in benchmark (mouse) | 33 | ||||||||
| Aligned words | 502 | ||||||||
| TF family | Ets-related factors {3.5.2} | ||||||||
| TF subfamily | Ets-like factors {3.5.2.1} | ||||||||
| HGNC | HGNC:3488 | ||||||||
| EntrezGene | GeneID:2113 (SSTAR profile) | ||||||||
| UniProt ID | ETS1_HUMAN | ||||||||
| UniProt AC | P14921 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | ETS1 expression | ||||||||
| ReMap ChIP-seq dataset list | ETS1 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 46.0 | 19.0 | 41.0 | 6.0 | 44.0 | 29.0 | 28.0 | 24.0 | 78.0 | 25.0 | 73.0 | 17.0 | 15.0 | 19.0 | 23.0 | 11.0 |
| 02 | 62.0 | 36.0 | 75.0 | 10.0 | 13.0 | 28.0 | 34.0 | 17.0 | 46.0 | 31.0 | 75.0 | 13.0 | 9.0 | 8.0 | 32.0 | 9.0 |
| 03 | 69.0 | 7.0 | 53.0 | 1.0 | 62.0 | 10.0 | 27.0 | 4.0 | 90.0 | 12.0 | 108.0 | 6.0 | 13.0 | 11.0 | 23.0 | 2.0 |
| 04 | 26.0 | 161.0 | 46.0 | 1.0 | 1.0 | 34.0 | 5.0 | 0.0 | 19.0 | 133.0 | 55.0 | 4.0 | 1.0 | 8.0 | 4.0 | 0.0 |
| 05 | 36.0 | 11.0 | 0.0 | 0.0 | 203.0 | 125.0 | 8.0 | 0.0 | 52.0 | 56.0 | 2.0 | 0.0 | 4.0 | 1.0 | 0.0 | 0.0 |
| 06 | 0.0 | 0.0 | 295.0 | 0.0 | 1.0 | 0.0 | 192.0 | 0.0 | 0.0 | 0.0 | 8.0 | 2.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 07 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 494.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 |
| 08 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 491.0 | 3.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 09 | 400.0 | 3.0 | 2.0 | 87.0 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 |
| 10 | 91.0 | 2.0 | 312.0 | 1.0 | 0.0 | 0.0 | 3.0 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 | 8.0 | 0.0 | 78.0 | 1.0 |
| 11 | 6.0 | 26.0 | 4.0 | 64.0 | 0.0 | 2.0 | 0.0 | 1.0 | 25.0 | 78.0 | 14.0 | 276.0 | 1.0 | 0.0 | 1.0 | 0.0 |
| 12 | 7.0 | 3.0 | 21.0 | 1.0 | 22.0 | 11.0 | 51.0 | 22.0 | 6.0 | 1.0 | 9.0 | 3.0 | 31.0 | 28.0 | 251.0 | 31.0 |
| 13 | 11.0 | 10.0 | 39.0 | 6.0 | 12.0 | 10.0 | 9.0 | 12.0 | 42.0 | 36.0 | 228.0 | 26.0 | 7.0 | 12.0 | 34.0 | 4.0 |
| 14 | 8.0 | 27.0 | 31.0 | 6.0 | 12.0 | 16.0 | 18.0 | 22.0 | 58.0 | 74.0 | 94.0 | 84.0 | 4.0 | 20.0 | 18.0 | 6.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 0.387 | -0.486 | 0.273 | -1.596 | 0.343 | -0.07 | -0.104 | -0.256 | 0.911 | -0.216 | 0.845 | -0.595 | -0.717 | -0.486 | -0.298 | -1.018 |
| 02 | 0.683 | 0.144 | 0.872 | -1.11 | -0.856 | -0.104 | 0.087 | -0.595 | 0.387 | -0.004 | 0.872 | -0.856 | -1.211 | -1.324 | 0.027 | -1.211 |
| 03 | 0.789 | -1.451 | 0.527 | -3.122 | 0.683 | -1.11 | -0.14 | -1.971 | 1.054 | -0.934 | 1.235 | -1.596 | -0.856 | -1.018 | -0.298 | -2.58 |
| 04 | -0.177 | 1.633 | 0.387 | -3.122 | -3.122 | 0.087 | -1.766 | -4.397 | -0.486 | 1.443 | 0.564 | -1.971 | -3.122 | -1.324 | -1.971 | -4.397 |
| 05 | 0.144 | -1.018 | -4.397 | -4.397 | 1.865 | 1.381 | -1.324 | -4.397 | 0.508 | 0.582 | -2.58 | -4.397 | -1.971 | -3.122 | -4.397 | -4.397 |
| 06 | -4.397 | -4.397 | 2.238 | -4.397 | -3.122 | -4.397 | 1.809 | -4.397 | -4.397 | -4.397 | -1.324 | -2.58 | -4.397 | -4.397 | -4.397 | -4.397 |
| 07 | -4.397 | -4.397 | -3.122 | -4.397 | -4.397 | -4.397 | -4.397 | -4.397 | -3.122 | -4.397 | 2.753 | -4.397 | -4.397 | -4.397 | -2.58 | -4.397 |
| 08 | -3.122 | -4.397 | -4.397 | -4.397 | -4.397 | -4.397 | -4.397 | -4.397 | 2.747 | -2.23 | -4.397 | -2.23 | -4.397 | -4.397 | -4.397 | -4.397 |
| 09 | 2.542 | -2.23 | -2.58 | 1.02 | -2.23 | -4.397 | -4.397 | -4.397 | -4.397 | -4.397 | -4.397 | -4.397 | -2.23 | -4.397 | -4.397 | -4.397 |
| 10 | 1.065 | -2.58 | 2.294 | -3.122 | -4.397 | -4.397 | -2.23 | -4.397 | -3.122 | -3.122 | -4.397 | -4.397 | -1.324 | -4.397 | 0.911 | -3.122 |
| 11 | -1.596 | -0.177 | -1.971 | 0.715 | -4.397 | -2.58 | -4.397 | -3.122 | -0.216 | 0.911 | -0.784 | 2.171 | -3.122 | -4.397 | -3.122 | -4.397 |
| 12 | -1.451 | -2.23 | -0.388 | -3.122 | -0.342 | -1.018 | 0.489 | -0.342 | -1.596 | -3.122 | -1.211 | -2.23 | -0.004 | -0.104 | 2.077 | -0.004 |
| 13 | -1.018 | -1.11 | 0.223 | -1.596 | -0.934 | -1.11 | -1.211 | -0.934 | 0.296 | 0.144 | 1.981 | -0.177 | -1.451 | -0.934 | 0.087 | -1.971 |
| 14 | -1.324 | -0.14 | -0.004 | -1.596 | -0.934 | -0.654 | -0.539 | -0.342 | 0.617 | 0.859 | 1.097 | 0.985 | -1.971 | -0.435 | -0.539 | -1.596 |