Model info
| Transcription factor | FOXH1 (GeneCards) | ||||||||
| Model | FOXH1_HUMAN.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 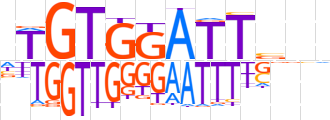 | ||||||||
| LOGO (reverse complement) | 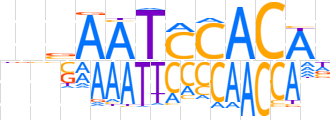 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 12 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | nTGTGGATTbnv | ||||||||
| Best auROC (human) | 0.982 | ||||||||
| Best auROC (mouse) | |||||||||
| Peak sets in benchmark (human) | 7 | ||||||||
| Peak sets in benchmark (mouse) | |||||||||
| Aligned words | 502 | ||||||||
| TF family | Forkhead box (FOX) factors {3.3.1} | ||||||||
| TF subfamily | FOXH {3.3.1.8} | ||||||||
| HGNC | HGNC:3814 | ||||||||
| EntrezGene | GeneID:8928 (SSTAR profile) | ||||||||
| UniProt ID | FOXH1_HUMAN | ||||||||
| UniProt AC | O75593 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | FOXH1 expression | ||||||||
| ReMap ChIP-seq dataset list | FOXH1 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 4.0 | 9.0 | 4.0 | 126.0 | 28.0 | 3.0 | 2.0 | 85.0 | 4.0 | 0.0 | 2.0 | 83.0 | 9.0 | 2.0 | 18.0 | 120.0 |
| 02 | 1.0 | 0.0 | 44.0 | 0.0 | 0.0 | 0.0 | 14.0 | 0.0 | 3.0 | 0.0 | 23.0 | 0.0 | 1.0 | 0.0 | 413.0 | 0.0 |
| 03 | 0.0 | 0.0 | 0.0 | 5.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 8.0 | 6.0 | 480.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 04 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 7.0 | 1.0 | 0.0 | 0.0 | 5.0 | 1.0 | 5.0 | 0.0 | 419.0 | 61.0 |
| 05 | 0.0 | 0.0 | 5.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 318.0 | 112.0 | 0.0 | 0.0 | 53.0 | 10.0 |
| 06 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 357.0 | 0.0 | 0.0 | 19.0 | 118.0 | 1.0 | 2.0 | 1.0 |
| 07 | 19.0 | 8.0 | 10.0 | 439.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 | 13.0 | 7.0 |
| 08 | 1.0 | 1.0 | 0.0 | 17.0 | 0.0 | 0.0 | 0.0 | 8.0 | 1.0 | 3.0 | 0.0 | 20.0 | 0.0 | 29.0 | 2.0 | 417.0 |
| 09 | 1.0 | 1.0 | 0.0 | 0.0 | 7.0 | 12.0 | 1.0 | 13.0 | 0.0 | 1.0 | 0.0 | 1.0 | 48.0 | 178.0 | 192.0 | 44.0 |
| 10 | 9.0 | 10.0 | 33.0 | 4.0 | 72.0 | 67.0 | 14.0 | 39.0 | 47.0 | 39.0 | 84.0 | 23.0 | 7.0 | 15.0 | 32.0 | 4.0 |
| 11 | 30.0 | 27.0 | 69.0 | 9.0 | 87.0 | 17.0 | 7.0 | 20.0 | 56.0 | 35.0 | 56.0 | 16.0 | 11.0 | 8.0 | 48.0 | 3.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -1.973 | -1.213 | -1.973 | 1.387 | -0.106 | -2.232 | -2.582 | 0.995 | -1.973 | -4.398 | -2.582 | 0.971 | -1.213 | -2.582 | -0.541 | 1.338 |
| 02 | -3.124 | -4.398 | 0.341 | -4.398 | -4.398 | -4.398 | -0.786 | -4.398 | -2.232 | -4.398 | -0.3 | -4.398 | -3.124 | -4.398 | 2.572 | -4.398 |
| 03 | -4.398 | -4.398 | -4.398 | -1.768 | -4.398 | -4.398 | -4.398 | -4.398 | -4.398 | -1.326 | -1.598 | 2.722 | -4.398 | -4.398 | -4.398 | -4.398 |
| 04 | -4.398 | -4.398 | -4.398 | -4.398 | -4.398 | -4.398 | -1.452 | -3.124 | -4.398 | -4.398 | -1.768 | -3.124 | -1.768 | -4.398 | 2.586 | 0.665 |
| 05 | -4.398 | -4.398 | -1.768 | -4.398 | -4.398 | -4.398 | -4.398 | -4.398 | -4.398 | -3.124 | 2.311 | 1.27 | -4.398 | -4.398 | 0.525 | -1.112 |
| 06 | -4.398 | -4.398 | -4.398 | -4.398 | -3.124 | -4.398 | -4.398 | -4.398 | 2.426 | -4.398 | -4.398 | -0.488 | 1.322 | -3.124 | -2.582 | -3.124 |
| 07 | -0.488 | -1.326 | -1.112 | 2.633 | -4.398 | -4.398 | -4.398 | -3.124 | -4.398 | -4.398 | -3.124 | -3.124 | -4.398 | -4.398 | -0.858 | -1.452 |
| 08 | -3.124 | -3.124 | -4.398 | -0.597 | -4.398 | -4.398 | -4.398 | -1.326 | -3.124 | -2.232 | -4.398 | -0.437 | -4.398 | -0.072 | -2.582 | 2.582 |
| 09 | -3.124 | -3.124 | -4.398 | -4.398 | -1.452 | -0.936 | -3.124 | -0.858 | -4.398 | -3.124 | -4.398 | -3.124 | 0.427 | 1.732 | 1.807 | 0.341 |
| 10 | -1.213 | -1.112 | 0.056 | -1.973 | 0.83 | 0.758 | -0.786 | 0.221 | 0.406 | 0.221 | 0.983 | -0.3 | -1.452 | -0.719 | 0.025 | -1.973 |
| 11 | -0.038 | -0.142 | 0.787 | -1.213 | 1.018 | -0.597 | -1.452 | -0.437 | 0.58 | 0.114 | 0.58 | -0.656 | -1.02 | -1.326 | 0.427 | -2.232 |