Model info
| Transcription factor | Hsf1 | ||||||||
| Model | HSF1_MOUSE.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 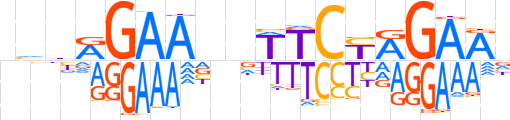 | ||||||||
| LOGO (reverse complement) |  | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 18 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | nvhRGAAnnWTCYRGAAn | ||||||||
| Best auROC (human) | 0.996 | ||||||||
| Best auROC (mouse) | 0.992 | ||||||||
| Peak sets in benchmark (human) | 56 | ||||||||
| Peak sets in benchmark (mouse) | 32 | ||||||||
| Aligned words | 505 | ||||||||
| TF family | HSF factors {3.4.1} | ||||||||
| TF subfamily | HSF1 (HSTF1) {3.4.1.0.1} | ||||||||
| MGI | MGI:96238 | ||||||||
| EntrezGene | GeneID:15499 (SSTAR profile) | ||||||||
| UniProt ID | HSF1_MOUSE | ||||||||
| UniProt AC | P38532 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | Hsf1 expression | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 26.0 | 42.0 | 19.0 | 13.0 | 53.0 | 52.0 | 11.0 | 16.0 | 30.0 | 31.0 | 36.0 | 12.0 | 19.0 | 93.0 | 28.0 | 19.0 |
| 02 | 25.0 | 36.0 | 32.0 | 35.0 | 27.0 | 77.0 | 14.0 | 100.0 | 26.0 | 15.0 | 21.0 | 32.0 | 4.0 | 17.0 | 14.0 | 25.0 |
| 03 | 47.0 | 1.0 | 32.0 | 2.0 | 114.0 | 6.0 | 21.0 | 4.0 | 53.0 | 2.0 | 23.0 | 3.0 | 86.0 | 5.0 | 99.0 | 2.0 |
| 04 | 0.0 | 1.0 | 299.0 | 0.0 | 0.0 | 0.0 | 14.0 | 0.0 | 1.0 | 0.0 | 173.0 | 1.0 | 0.0 | 0.0 | 11.0 | 0.0 |
| 05 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 473.0 | 15.0 | 4.0 | 5.0 | 1.0 | 0.0 | 0.0 | 0.0 |
| 06 | 449.0 | 4.0 | 10.0 | 12.0 | 12.0 | 1.0 | 1.0 | 1.0 | 3.0 | 0.0 | 1.0 | 0.0 | 1.0 | 1.0 | 4.0 | 0.0 |
| 07 | 132.0 | 115.0 | 122.0 | 96.0 | 3.0 | 3.0 | 0.0 | 0.0 | 6.0 | 2.0 | 2.0 | 6.0 | 2.0 | 6.0 | 4.0 | 1.0 |
| 08 | 29.0 | 32.0 | 69.0 | 13.0 | 44.0 | 39.0 | 4.0 | 39.0 | 26.0 | 28.0 | 51.0 | 23.0 | 11.0 | 14.0 | 61.0 | 17.0 |
| 09 | 11.0 | 7.0 | 4.0 | 88.0 | 18.0 | 5.0 | 1.0 | 89.0 | 12.0 | 9.0 | 10.0 | 154.0 | 3.0 | 17.0 | 3.0 | 69.0 |
| 10 | 7.0 | 1.0 | 5.0 | 31.0 | 1.0 | 0.0 | 2.0 | 35.0 | 3.0 | 1.0 | 3.0 | 11.0 | 19.0 | 13.0 | 23.0 | 345.0 |
| 11 | 1.0 | 29.0 | 0.0 | 0.0 | 1.0 | 13.0 | 0.0 | 1.0 | 0.0 | 32.0 | 0.0 | 1.0 | 1.0 | 415.0 | 6.0 | 0.0 |
| 12 | 0.0 | 1.0 | 0.0 | 2.0 | 8.0 | 158.0 | 18.0 | 305.0 | 0.0 | 0.0 | 4.0 | 2.0 | 0.0 | 0.0 | 0.0 | 2.0 |
| 13 | 3.0 | 1.0 | 4.0 | 0.0 | 143.0 | 2.0 | 14.0 | 0.0 | 14.0 | 2.0 | 5.0 | 1.0 | 181.0 | 3.0 | 125.0 | 2.0 |
| 14 | 0.0 | 0.0 | 339.0 | 2.0 | 1.0 | 0.0 | 7.0 | 0.0 | 0.0 | 0.0 | 147.0 | 1.0 | 0.0 | 0.0 | 3.0 | 0.0 |
| 15 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 456.0 | 10.0 | 16.0 | 14.0 | 1.0 | 0.0 | 2.0 | 0.0 |
| 16 | 386.0 | 13.0 | 41.0 | 18.0 | 9.0 | 0.0 | 0.0 | 1.0 | 5.0 | 7.0 | 5.0 | 1.0 | 7.0 | 1.0 | 4.0 | 2.0 |
| 17 | 105.0 | 87.0 | 135.0 | 80.0 | 4.0 | 3.0 | 2.0 | 12.0 | 11.0 | 12.0 | 17.0 | 10.0 | 7.0 | 6.0 | 6.0 | 3.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -0.181 | 0.293 | -0.49 | -0.86 | 0.523 | 0.504 | -1.022 | -0.658 | -0.04 | -0.008 | 0.14 | -0.938 | -0.49 | 1.082 | -0.108 | -0.49 |
| 02 | -0.22 | 0.14 | 0.023 | 0.112 | -0.144 | 0.894 | -0.788 | 1.155 | -0.181 | -0.721 | -0.392 | 0.023 | -1.975 | -0.599 | -0.788 | -0.22 |
| 03 | 0.404 | -3.126 | 0.023 | -2.584 | 1.285 | -1.6 | -0.392 | -1.975 | 0.523 | -2.584 | -0.302 | -2.234 | 1.004 | -1.77 | 1.145 | -2.584 |
| 04 | -4.4 | -3.126 | 2.247 | -4.4 | -4.4 | -4.4 | -0.788 | -4.4 | -3.126 | -4.4 | 1.701 | -3.126 | -4.4 | -4.4 | -1.022 | -4.4 |
| 05 | -3.126 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -3.126 | 2.706 | -0.721 | -1.975 | -1.77 | -3.126 | -4.4 | -4.4 | -4.4 |
| 06 | 2.654 | -1.975 | -1.114 | -0.938 | -0.938 | -3.126 | -3.126 | -3.126 | -2.234 | -4.4 | -3.126 | -4.4 | -3.126 | -3.126 | -1.975 | -4.4 |
| 07 | 1.431 | 1.294 | 1.353 | 1.114 | -2.234 | -2.234 | -4.4 | -4.4 | -1.6 | -2.584 | -2.584 | -1.6 | -2.584 | -1.6 | -1.975 | -3.126 |
| 08 | -0.074 | 0.023 | 0.785 | -0.86 | 0.339 | 0.219 | -1.975 | 0.219 | -0.181 | -0.108 | 0.485 | -0.302 | -1.022 | -0.788 | 0.663 | -0.599 |
| 09 | -1.022 | -1.454 | -1.975 | 1.027 | -0.543 | -1.77 | -3.126 | 1.039 | -0.938 | -1.215 | -1.114 | 1.585 | -2.234 | -0.599 | -2.234 | 0.785 |
| 10 | -1.454 | -3.126 | -1.77 | -0.008 | -3.126 | -4.4 | -2.584 | 0.112 | -2.234 | -3.126 | -2.234 | -1.022 | -0.49 | -0.86 | -0.302 | 2.39 |
| 11 | -3.126 | -0.074 | -4.4 | -4.4 | -3.126 | -0.86 | -4.4 | -3.126 | -4.4 | 0.023 | -4.4 | -3.126 | -3.126 | 2.575 | -1.6 | -4.4 |
| 12 | -4.4 | -3.126 | -4.4 | -2.584 | -1.328 | 1.611 | -0.543 | 2.267 | -4.4 | -4.4 | -1.975 | -2.584 | -4.4 | -4.4 | -4.4 | -2.584 |
| 13 | -2.234 | -3.126 | -1.975 | -4.4 | 1.511 | -2.584 | -0.788 | -4.4 | -0.788 | -2.584 | -1.77 | -3.126 | 1.746 | -2.234 | 1.377 | -2.584 |
| 14 | -4.4 | -4.4 | 2.373 | -2.584 | -3.126 | -4.4 | -1.454 | -4.4 | -4.4 | -4.4 | 1.539 | -3.126 | -4.4 | -4.4 | -2.234 | -4.4 |
| 15 | -3.126 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | 2.669 | -1.114 | -0.658 | -0.788 | -3.126 | -4.4 | -2.584 | -4.4 |
| 16 | 2.502 | -0.86 | 0.269 | -0.543 | -1.215 | -4.4 | -4.4 | -3.126 | -1.77 | -1.454 | -1.77 | -3.126 | -1.454 | -3.126 | -1.975 | -2.584 |
| 17 | 1.203 | 1.016 | 1.454 | 0.932 | -1.975 | -2.234 | -2.584 | -0.938 | -1.022 | -0.938 | -0.599 | -1.114 | -1.454 | -1.6 | -1.6 | -2.234 |