Model info
| Transcription factor | Jun | ||||||||
| Model | JUN_MOUSE.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 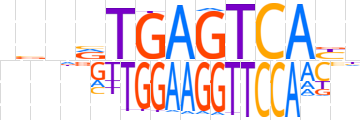 | ||||||||
| LOGO (reverse complement) |  | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 13 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | nbnRTGAGTCAYn | ||||||||
| Best auROC (human) | 0.944 | ||||||||
| Best auROC (mouse) | 0.94 | ||||||||
| Peak sets in benchmark (human) | 56 | ||||||||
| Peak sets in benchmark (mouse) | 39 | ||||||||
| Aligned words | 481 | ||||||||
| TF family | Jun-related factors {1.1.1} | ||||||||
| TF subfamily | Jun factors {1.1.1.1} | ||||||||
| MGI | MGI:96646 | ||||||||
| EntrezGene | GeneID:16476 (SSTAR profile) | ||||||||
| UniProt ID | JUN_MOUSE | ||||||||
| UniProt AC | P05627 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | Jun expression | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 21.0 | 18.0 | 71.0 | 10.0 | 14.0 | 41.0 | 15.0 | 37.0 | 21.0 | 41.0 | 63.0 | 24.0 | 7.0 | 17.0 | 56.0 | 20.0 |
| 02 | 22.0 | 9.0 | 23.0 | 9.0 | 32.0 | 31.0 | 11.0 | 43.0 | 54.0 | 49.0 | 76.0 | 26.0 | 12.0 | 15.0 | 39.0 | 25.0 |
| 03 | 15.0 | 10.0 | 90.0 | 5.0 | 44.0 | 27.0 | 27.0 | 6.0 | 35.0 | 39.0 | 73.0 | 2.0 | 11.0 | 21.0 | 70.0 | 1.0 |
| 04 | 1.0 | 2.0 | 1.0 | 101.0 | 2.0 | 1.0 | 0.0 | 94.0 | 2.0 | 2.0 | 1.0 | 255.0 | 1.0 | 0.0 | 2.0 | 11.0 |
| 05 | 0.0 | 0.0 | 6.0 | 0.0 | 0.0 | 2.0 | 1.0 | 2.0 | 0.0 | 0.0 | 4.0 | 0.0 | 6.0 | 1.0 | 438.0 | 16.0 |
| 06 | 4.0 | 1.0 | 0.0 | 1.0 | 3.0 | 0.0 | 0.0 | 0.0 | 441.0 | 2.0 | 2.0 | 4.0 | 18.0 | 0.0 | 0.0 | 0.0 |
| 07 | 25.0 | 0.0 | 441.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 5.0 | 0.0 |
| 08 | 0.0 | 0.0 | 0.0 | 26.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 5.0 | 2.0 | 442.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 09 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 4.0 | 0.0 | 1.0 | 0.0 | 2.0 | 0.0 | 0.0 | 1.0 | 463.0 | 3.0 | 1.0 |
| 10 | 1.0 | 0.0 | 0.0 | 0.0 | 464.0 | 5.0 | 0.0 | 1.0 | 2.0 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 | 0.0 | 1.0 |
| 11 | 15.0 | 257.0 | 84.0 | 112.0 | 1.0 | 1.0 | 0.0 | 3.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 1.0 |
| 12 | 1.0 | 4.0 | 9.0 | 3.0 | 74.0 | 81.0 | 18.0 | 85.0 | 39.0 | 24.0 | 15.0 | 7.0 | 18.0 | 34.0 | 35.0 | 29.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -0.343 | -0.494 | 0.862 | -1.065 | -0.739 | 0.317 | -0.672 | 0.216 | -0.343 | 0.317 | 0.744 | -0.212 | -1.406 | -0.55 | 0.627 | -0.391 |
| 02 | -0.297 | -1.167 | -0.254 | -1.167 | 0.072 | 0.041 | -0.973 | 0.364 | 0.59 | 0.494 | 0.93 | -0.133 | -0.889 | -0.672 | 0.268 | -0.172 |
| 03 | -0.672 | -1.065 | 1.098 | -1.722 | 0.387 | -0.096 | -0.096 | -1.552 | 0.161 | 0.268 | 0.89 | -2.536 | -0.973 | -0.343 | 0.848 | -3.08 |
| 04 | -3.08 | -2.536 | -3.08 | 1.213 | -2.536 | -3.08 | -4.359 | 1.142 | -2.536 | -2.536 | -3.08 | 2.137 | -3.08 | -4.359 | -2.536 | -0.973 |
| 05 | -4.359 | -4.359 | -1.552 | -4.359 | -4.359 | -2.536 | -3.08 | -2.536 | -4.359 | -4.359 | -1.927 | -4.359 | -1.552 | -3.08 | 2.677 | -0.609 |
| 06 | -1.927 | -3.08 | -4.359 | -3.08 | -2.186 | -4.359 | -4.359 | -4.359 | 2.684 | -2.536 | -2.536 | -1.927 | -0.494 | -4.359 | -4.359 | -4.359 |
| 07 | -0.172 | -4.359 | 2.684 | -4.359 | -4.359 | -4.359 | -2.186 | -4.359 | -3.08 | -4.359 | -3.08 | -4.359 | -4.359 | -4.359 | -1.722 | -4.359 |
| 08 | -4.359 | -4.359 | -4.359 | -0.133 | -4.359 | -4.359 | -4.359 | -4.359 | -3.08 | -1.722 | -2.536 | 2.686 | -4.359 | -4.359 | -4.359 | -4.359 |
| 09 | -4.359 | -3.08 | -4.359 | -4.359 | -4.359 | -1.927 | -4.359 | -3.08 | -4.359 | -2.536 | -4.359 | -4.359 | -3.08 | 2.733 | -2.186 | -3.08 |
| 10 | -3.08 | -4.359 | -4.359 | -4.359 | 2.735 | -1.722 | -4.359 | -3.08 | -2.536 | -4.359 | -3.08 | -4.359 | -3.08 | -4.359 | -4.359 | -3.08 |
| 11 | -0.672 | 2.145 | 1.03 | 1.316 | -3.08 | -3.08 | -4.359 | -2.186 | -3.08 | -4.359 | -4.359 | -4.359 | -4.359 | -4.359 | -3.08 | -3.08 |
| 12 | -3.08 | -1.927 | -1.167 | -2.186 | 0.904 | 0.993 | -0.494 | 1.041 | 0.268 | -0.212 | -0.672 | -1.406 | -0.494 | 0.132 | 0.161 | -0.025 |