Model info
| Transcription factor | KLF9 (GeneCards) | ||||||||
| Model | KLF9_HUMAN.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 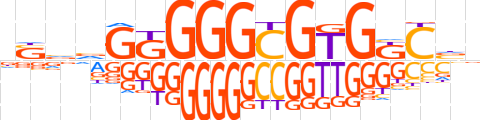 | ||||||||
| LOGO (reverse complement) | 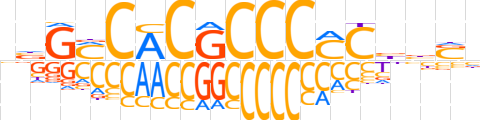 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 17 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | nSvvGKGGGCGTGKCvb | ||||||||
| Best auROC (human) | 0.957 | ||||||||
| Best auROC (mouse) | |||||||||
| Peak sets in benchmark (human) | 3 | ||||||||
| Peak sets in benchmark (mouse) | |||||||||
| Aligned words | 252 | ||||||||
| TF family | Three-zinc finger Krüppel-related factors {2.3.1} | ||||||||
| TF subfamily | Krüppel-like factors {2.3.1.2} | ||||||||
| HGNC | HGNC:1123 | ||||||||
| EntrezGene | GeneID:687 (SSTAR profile) | ||||||||
| UniProt ID | KLF9_HUMAN | ||||||||
| UniProt AC | Q13886 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | KLF9 expression | ||||||||
| ReMap ChIP-seq dataset list | KLF9 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 1.0 | 12.0 | 26.0 | 5.0 | 5.0 | 14.0 | 29.0 | 3.0 | 5.0 | 10.0 | 62.0 | 11.0 | 4.0 | 10.0 | 35.0 | 4.0 |
| 02 | 6.0 | 3.0 | 6.0 | 0.0 | 7.0 | 13.0 | 20.0 | 6.0 | 43.0 | 26.0 | 71.0 | 12.0 | 3.0 | 7.0 | 8.0 | 5.0 |
| 03 | 29.0 | 7.0 | 19.0 | 4.0 | 21.0 | 10.0 | 11.0 | 7.0 | 57.0 | 17.0 | 19.0 | 12.0 | 7.0 | 1.0 | 11.0 | 4.0 |
| 04 | 14.0 | 0.0 | 100.0 | 0.0 | 4.0 | 2.0 | 27.0 | 2.0 | 9.0 | 1.0 | 47.0 | 3.0 | 1.0 | 0.0 | 26.0 | 0.0 |
| 05 | 1.0 | 0.0 | 22.0 | 5.0 | 0.0 | 0.0 | 1.0 | 2.0 | 3.0 | 0.0 | 129.0 | 68.0 | 0.0 | 0.0 | 4.0 | 1.0 |
| 06 | 0.0 | 0.0 | 4.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 156.0 | 0.0 | 0.0 | 0.0 | 76.0 | 0.0 |
| 07 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 235.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 08 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 1.0 | 233.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 |
| 09 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 1.0 | 1.0 | 187.0 | 0.0 | 46.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 10 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 187.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 48.0 | 0.0 |
| 11 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 58.0 | 177.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 12 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 56.0 | 2.0 | 0.0 | 0.0 | 176.0 | 1.0 |
| 13 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 14.0 | 24.0 | 160.0 | 35.0 | 0.0 | 0.0 | 3.0 | 0.0 |
| 14 | 0.0 | 12.0 | 1.0 | 1.0 | 0.0 | 21.0 | 1.0 | 2.0 | 1.0 | 135.0 | 6.0 | 21.0 | 1.0 | 34.0 | 0.0 | 0.0 |
| 15 | 1.0 | 0.0 | 0.0 | 1.0 | 31.0 | 109.0 | 38.0 | 24.0 | 2.0 | 3.0 | 2.0 | 1.0 | 0.0 | 12.0 | 6.0 | 6.0 |
| 16 | 2.0 | 10.0 | 21.0 | 1.0 | 21.0 | 27.0 | 46.0 | 30.0 | 8.0 | 18.0 | 14.0 | 6.0 | 3.0 | 12.0 | 14.0 | 3.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -2.42 | -0.201 | 0.557 | -1.039 | -1.039 | -0.051 | 0.665 | -1.508 | -1.039 | -0.378 | 1.418 | -0.286 | -1.246 | -0.378 | 0.851 | -1.246 |
| 02 | -0.867 | -1.508 | -0.867 | -3.789 | -0.721 | -0.123 | 0.299 | -0.867 | 1.055 | 0.557 | 1.553 | -0.201 | -1.508 | -0.721 | -0.593 | -1.039 |
| 03 | 0.665 | -0.721 | 0.248 | -1.246 | 0.347 | -0.378 | -0.286 | -0.721 | 1.335 | 0.139 | 0.248 | -0.201 | -0.721 | -2.42 | -0.286 | -1.246 |
| 04 | -0.051 | -3.789 | 1.894 | -3.789 | -1.246 | -1.863 | 0.594 | -1.863 | -0.48 | -2.42 | 1.143 | -1.508 | -2.42 | -3.789 | 0.557 | -3.789 |
| 05 | -2.42 | -3.789 | 0.392 | -1.039 | -3.789 | -3.789 | -2.42 | -1.863 | -1.508 | -3.789 | 2.148 | 1.51 | -3.789 | -3.789 | -1.246 | -2.42 |
| 06 | -3.789 | -3.789 | -1.246 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | 2.338 | -3.789 | -3.789 | -3.789 | 1.621 | -3.789 |
| 07 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | 2.747 | -2.42 | -3.789 | -3.789 | -3.789 | -3.789 |
| 08 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -2.42 | -2.42 | 2.738 | -3.789 | -3.789 | -3.789 | -2.42 | -3.789 |
| 09 | -3.789 | -3.789 | -3.789 | -2.42 | -3.789 | -3.789 | -3.789 | -2.42 | -2.42 | 2.519 | -3.789 | 1.122 | -3.789 | -3.789 | -3.789 | -3.789 |
| 10 | -3.789 | -3.789 | -2.42 | -3.789 | -3.789 | -3.789 | 2.519 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | 1.164 | -3.789 |
| 11 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -2.42 | 1.352 | 2.464 | -3.789 | -3.789 | -3.789 | -3.789 |
| 12 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -2.42 | -3.789 | -3.789 | -3.789 | 1.317 | -1.863 | -3.789 | -3.789 | 2.458 | -2.42 |
| 13 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -3.789 | -0.051 | 0.478 | 2.363 | 0.851 | -3.789 | -3.789 | -1.508 | -3.789 |
| 14 | -3.789 | -0.201 | -2.42 | -2.42 | -3.789 | 0.347 | -2.42 | -1.863 | -2.42 | 2.194 | -0.867 | 0.347 | -2.42 | 0.822 | -3.789 | -3.789 |
| 15 | -2.42 | -3.789 | -3.789 | -2.42 | 0.731 | 1.98 | 0.932 | 0.478 | -1.863 | -1.508 | -1.863 | -2.42 | -3.789 | -0.201 | -0.867 | -0.867 |
| 16 | -1.863 | -0.378 | 0.347 | -2.42 | 0.347 | 0.594 | 1.122 | 0.698 | -0.593 | 0.195 | -0.051 | -0.867 | -1.508 | -0.201 | -0.051 | -1.508 |