Model info
| Transcription factor | ZNF146 (GeneCards) | ||||||||
| Model | OZF_HUMAN.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 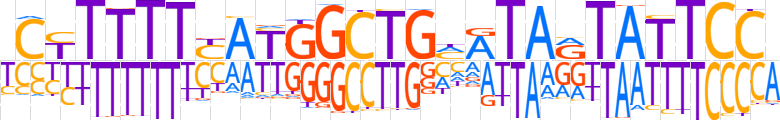 | ||||||||
| LOGO (reverse complement) |  | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 27 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | YCYTTTTYATGGCTGMATARTATTCCA | ||||||||
| Best auROC (human) | 0.992 | ||||||||
| Best auROC (mouse) | |||||||||
| Peak sets in benchmark (human) | 4 | ||||||||
| Peak sets in benchmark (mouse) | |||||||||
| Aligned words | 453 | ||||||||
| TF family | More than 3 adjacent zinc finger factors {2.3.3} | ||||||||
| TF subfamily | ZNF146-like factors {2.3.3.55} | ||||||||
| HGNC | HGNC:12931 | ||||||||
| EntrezGene | GeneID:7705 (SSTAR profile) | ||||||||
| UniProt ID | OZF_HUMAN | ||||||||
| UniProt AC | Q15072 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | ZNF146 expression | ||||||||
| ReMap ChIP-seq dataset list | ZNF146 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 0.0 | 1.0 | 0.0 | 1.0 | 18.0 | 103.0 | 1.0 | 3.0 | 3.0 | 8.0 | 0.0 | 0.0 | 8.0 | 297.0 | 3.0 | 4.0 |
| 02 | 1.0 | 5.0 | 0.0 | 23.0 | 6.0 | 175.0 | 0.0 | 228.0 | 0.0 | 2.0 | 0.0 | 2.0 | 0.0 | 3.0 | 0.0 | 5.0 |
| 03 | 0.0 | 0.0 | 0.0 | 7.0 | 0.0 | 0.0 | 0.0 | 185.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 258.0 |
| 04 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 7.0 | 10.0 | 433.0 |
| 05 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 7.0 | 0.0 | 0.0 | 0.0 | 10.0 | 0.0 | 1.0 | 1.0 | 431.0 |
| 06 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 1.0 | 3.0 | 0.0 | 2.0 | 443.0 |
| 07 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 1.0 | 14.0 | 292.0 | 23.0 | 116.0 |
| 08 | 12.0 | 1.0 | 0.0 | 1.0 | 276.0 | 10.0 | 2.0 | 8.0 | 21.0 | 0.0 | 1.0 | 1.0 | 105.0 | 7.0 | 5.0 | 0.0 |
| 09 | 23.0 | 29.0 | 14.0 | 348.0 | 4.0 | 2.0 | 0.0 | 12.0 | 0.0 | 0.0 | 0.0 | 8.0 | 0.0 | 4.0 | 0.0 | 6.0 |
| 10 | 0.0 | 0.0 | 27.0 | 0.0 | 13.0 | 0.0 | 12.0 | 10.0 | 0.0 | 0.0 | 12.0 | 2.0 | 2.0 | 0.0 | 339.0 | 33.0 |
| 11 | 0.0 | 1.0 | 14.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 8.0 | 0.0 | 382.0 | 0.0 | 1.0 | 0.0 | 44.0 | 0.0 |
| 12 | 0.0 | 8.0 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 417.0 | 7.0 | 16.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 13 | 0.0 | 0.0 | 0.0 | 0.0 | 6.0 | 15.0 | 0.0 | 405.0 | 0.0 | 0.0 | 0.0 | 7.0 | 1.0 | 1.0 | 0.0 | 15.0 |
| 14 | 0.0 | 0.0 | 7.0 | 0.0 | 2.0 | 0.0 | 13.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 11.0 | 4.0 | 411.0 | 1.0 |
| 15 | 10.0 | 1.0 | 0.0 | 2.0 | 2.0 | 1.0 | 0.0 | 1.0 | 143.0 | 205.0 | 1.0 | 82.0 | 2.0 | 0.0 | 0.0 | 0.0 |
| 16 | 85.0 | 8.0 | 64.0 | 0.0 | 188.0 | 0.0 | 19.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 56.0 | 1.0 | 28.0 | 0.0 |
| 17 | 0.0 | 0.0 | 0.0 | 329.0 | 0.0 | 0.0 | 0.0 | 9.0 | 0.0 | 0.0 | 0.0 | 112.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 18 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 440.0 | 3.0 | 4.0 | 3.0 |
| 19 | 141.0 | 27.0 | 268.0 | 4.0 | 1.0 | 0.0 | 2.0 | 0.0 | 1.0 | 0.0 | 3.0 | 0.0 | 1.0 | 0.0 | 2.0 | 0.0 |
| 20 | 0.0 | 0.0 | 0.0 | 144.0 | 0.0 | 1.0 | 0.0 | 26.0 | 0.0 | 2.0 | 0.0 | 273.0 | 0.0 | 0.0 | 0.0 | 4.0 |
| 21 | 0.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 436.0 | 0.0 | 9.0 | 2.0 |
| 22 | 0.0 | 43.0 | 3.0 | 393.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 9.0 | 0.0 | 0.0 | 0.0 | 2.0 |
| 23 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 43.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 404.0 |
| 24 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 445.0 | 0.0 | 4.0 |
| 25 | 0.0 | 1.0 | 0.0 | 0.0 | 2.0 | 422.0 | 0.0 | 21.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 4.0 | 0.0 | 0.0 |
| 26 | 1.0 | 0.0 | 0.0 | 1.0 | 378.0 | 13.0 | 24.0 | 12.0 | 0.0 | 0.0 | 0.0 | 0.0 | 12.0 | 0.0 | 9.0 | 0.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -4.313 | -3.027 | -4.313 | -3.027 | -0.439 | 1.288 | -3.027 | -2.132 | -2.132 | -1.224 | -4.313 | -4.313 | -1.224 | 2.345 | -2.132 | -1.873 |
| 02 | -3.027 | -1.667 | -4.313 | -0.198 | -1.497 | 1.817 | -4.313 | 2.081 | -4.313 | -2.482 | -4.313 | -2.482 | -4.313 | -2.132 | -4.313 | -1.667 |
| 03 | -4.313 | -4.313 | -4.313 | -1.351 | -4.313 | -4.313 | -4.313 | 1.872 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | 2.204 |
| 04 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -1.351 | -1.01 | 2.721 |
| 05 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -1.351 | -4.313 | -4.313 | -4.313 | -1.01 | -4.313 | -3.027 | -3.027 | 2.717 |
| 06 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -3.027 | -4.313 | -4.313 | -4.313 | -3.027 | -2.132 | -4.313 | -2.482 | 2.744 |
| 07 | -4.313 | -2.132 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -3.027 | -4.313 | -3.027 | -0.684 | 2.328 | -0.198 | 1.407 |
| 08 | -0.834 | -3.027 | -4.313 | -3.027 | 2.272 | -1.01 | -2.482 | -1.224 | -0.288 | -4.313 | -3.027 | -3.027 | 1.307 | -1.351 | -1.667 | -4.313 |
| 09 | -0.198 | 0.03 | -0.684 | 2.503 | -1.873 | -2.482 | -4.313 | -0.834 | -4.313 | -4.313 | -4.313 | -1.224 | -4.313 | -1.873 | -4.313 | -1.497 |
| 10 | -4.313 | -4.313 | -0.04 | -4.313 | -0.756 | -4.313 | -0.834 | -1.01 | -4.313 | -4.313 | -0.834 | -2.482 | -2.482 | -4.313 | 2.477 | 0.158 |
| 11 | -4.313 | -3.027 | -0.684 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -1.224 | -4.313 | 2.596 | -4.313 | -3.027 | -4.313 | 0.443 | -4.313 |
| 12 | -4.313 | -1.224 | -4.313 | -3.027 | -4.313 | -3.027 | -4.313 | -4.313 | -4.313 | 2.684 | -1.351 | -0.554 | -4.313 | -4.313 | -4.313 | -4.313 |
| 13 | -4.313 | -4.313 | -4.313 | -4.313 | -1.497 | -0.617 | -4.313 | 2.655 | -4.313 | -4.313 | -4.313 | -1.351 | -3.027 | -3.027 | -4.313 | -0.617 |
| 14 | -4.313 | -4.313 | -1.351 | -4.313 | -2.482 | -4.313 | -0.756 | -3.027 | -4.313 | -4.313 | -4.313 | -4.313 | -0.918 | -1.873 | 2.669 | -3.027 |
| 15 | -1.01 | -3.027 | -4.313 | -2.482 | -2.482 | -3.027 | -4.313 | -3.027 | 1.615 | 1.975 | -3.027 | 1.061 | -2.482 | -4.313 | -4.313 | -4.313 |
| 16 | 1.097 | -1.224 | 0.815 | -4.313 | 1.888 | -4.313 | -0.386 | -4.313 | -4.313 | -4.313 | -3.027 | -4.313 | 0.682 | -3.027 | -0.004 | -4.313 |
| 17 | -4.313 | -4.313 | -4.313 | 2.447 | -4.313 | -4.313 | -4.313 | -1.111 | -4.313 | -4.313 | -4.313 | 1.372 | -4.313 | -4.313 | -4.313 | -4.313 |
| 18 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | 2.737 | -2.132 | -1.873 | -2.132 |
| 19 | 1.601 | -0.04 | 2.242 | -1.873 | -3.027 | -4.313 | -2.482 | -4.313 | -3.027 | -4.313 | -2.132 | -4.313 | -3.027 | -4.313 | -2.482 | -4.313 |
| 20 | -4.313 | -4.313 | -4.313 | 1.622 | -4.313 | -3.027 | -4.313 | -0.077 | -4.313 | -2.482 | -4.313 | 2.261 | -4.313 | -4.313 | -4.313 | -1.873 |
| 21 | -4.313 | -4.313 | -4.313 | -4.313 | -2.132 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | 2.728 | -4.313 | -1.111 | -2.482 |
| 22 | -4.313 | 0.42 | -2.132 | 2.625 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -1.111 | -4.313 | -4.313 | -4.313 | -2.482 |
| 23 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | 0.42 | -4.313 | -4.313 | -4.313 | -2.132 | -4.313 | -4.313 | -4.313 | 2.652 |
| 24 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -3.027 | 2.749 | -4.313 | -1.873 |
| 25 | -4.313 | -3.027 | -4.313 | -4.313 | -2.482 | 2.696 | -4.313 | -0.288 | -4.313 | -4.313 | -4.313 | -4.313 | -4.313 | -1.873 | -4.313 | -4.313 |
| 26 | -3.027 | -4.313 | -4.313 | -3.027 | 2.586 | -0.756 | -0.156 | -0.834 | -4.313 | -4.313 | -4.313 | -4.313 | -0.834 | -4.313 | -1.111 | -4.313 |