Model info
| Transcription factor | Prdm9 | ||||||||
| Model | PRDM9_MOUSE.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO |  | ||||||||
| LOGO (reverse complement) | 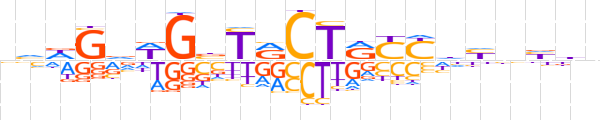 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 21 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | nhdnddRRYAGYAvCWbChbv | ||||||||
| Best auROC (human) | |||||||||
| Best auROC (mouse) | 0.91 | ||||||||
| Peak sets in benchmark (human) | |||||||||
| Peak sets in benchmark (mouse) | 4 | ||||||||
| Aligned words | 245 | ||||||||
| TF family | Factors with multiple dispersed zinc fingers {2.3.4} | ||||||||
| TF subfamily | unclassified {2.3.4.0} | ||||||||
| MGI | MGI:2384854 | ||||||||
| EntrezGene | |||||||||
| UniProt ID | PRDM9_MOUSE | ||||||||
| UniProt AC | Q96EQ9 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | Prdm9 expression | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 25.0 | 19.0 | 7.0 | 6.0 | 32.0 | 9.0 | 2.0 | 11.0 | 20.0 | 9.0 | 10.0 | 10.0 | 27.0 | 19.0 | 15.0 | 24.0 |
| 02 | 49.0 | 8.0 | 32.0 | 15.0 | 45.0 | 6.0 | 2.0 | 3.0 | 14.0 | 6.0 | 6.0 | 8.0 | 25.0 | 8.0 | 11.0 | 7.0 |
| 03 | 32.0 | 16.0 | 49.0 | 36.0 | 18.0 | 3.0 | 3.0 | 4.0 | 15.0 | 11.0 | 17.0 | 8.0 | 6.0 | 8.0 | 14.0 | 5.0 |
| 04 | 38.0 | 3.0 | 25.0 | 5.0 | 27.0 | 0.0 | 4.0 | 7.0 | 39.0 | 12.0 | 21.0 | 11.0 | 22.0 | 1.0 | 27.0 | 3.0 |
| 05 | 43.0 | 10.0 | 37.0 | 36.0 | 9.0 | 1.0 | 0.0 | 6.0 | 19.0 | 15.0 | 6.0 | 37.0 | 6.0 | 2.0 | 11.0 | 7.0 |
| 06 | 8.0 | 0.0 | 62.0 | 7.0 | 8.0 | 1.0 | 7.0 | 12.0 | 7.0 | 0.0 | 45.0 | 2.0 | 9.0 | 3.0 | 65.0 | 9.0 |
| 07 | 12.0 | 1.0 | 19.0 | 0.0 | 2.0 | 2.0 | 0.0 | 0.0 | 41.0 | 3.0 | 132.0 | 3.0 | 5.0 | 3.0 | 18.0 | 4.0 |
| 08 | 4.0 | 33.0 | 3.0 | 20.0 | 0.0 | 6.0 | 0.0 | 3.0 | 10.0 | 97.0 | 5.0 | 57.0 | 0.0 | 4.0 | 0.0 | 3.0 |
| 09 | 9.0 | 0.0 | 5.0 | 0.0 | 136.0 | 0.0 | 1.0 | 3.0 | 8.0 | 0.0 | 0.0 | 0.0 | 54.0 | 0.0 | 27.0 | 2.0 |
| 10 | 6.0 | 2.0 | 197.0 | 2.0 | 0.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 31.0 | 0.0 | 0.0 | 0.0 | 4.0 | 1.0 |
| 11 | 1.0 | 7.0 | 0.0 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 | 6.0 | 116.0 | 13.0 | 97.0 | 0.0 | 2.0 | 1.0 | 0.0 |
| 12 | 4.0 | 0.0 | 4.0 | 0.0 | 121.0 | 1.0 | 1.0 | 2.0 | 7.0 | 1.0 | 5.0 | 2.0 | 67.0 | 3.0 | 23.0 | 4.0 |
| 13 | 36.0 | 48.0 | 96.0 | 19.0 | 3.0 | 1.0 | 0.0 | 1.0 | 2.0 | 8.0 | 18.0 | 5.0 | 0.0 | 2.0 | 5.0 | 1.0 |
| 14 | 2.0 | 35.0 | 2.0 | 2.0 | 0.0 | 59.0 | 0.0 | 0.0 | 7.0 | 109.0 | 0.0 | 3.0 | 0.0 | 25.0 | 0.0 | 1.0 |
| 15 | 3.0 | 0.0 | 3.0 | 3.0 | 133.0 | 17.0 | 2.0 | 76.0 | 0.0 | 1.0 | 1.0 | 0.0 | 5.0 | 1.0 | 0.0 | 0.0 |
| 16 | 12.0 | 61.0 | 31.0 | 37.0 | 7.0 | 3.0 | 3.0 | 6.0 | 1.0 | 0.0 | 2.0 | 3.0 | 3.0 | 12.0 | 16.0 | 48.0 |
| 17 | 4.0 | 14.0 | 5.0 | 0.0 | 7.0 | 64.0 | 0.0 | 5.0 | 1.0 | 45.0 | 4.0 | 2.0 | 3.0 | 84.0 | 7.0 | 0.0 |
| 18 | 6.0 | 3.0 | 2.0 | 4.0 | 51.0 | 32.0 | 5.0 | 119.0 | 8.0 | 1.0 | 3.0 | 4.0 | 4.0 | 2.0 | 1.0 | 0.0 |
| 19 | 8.0 | 16.0 | 28.0 | 17.0 | 13.0 | 7.0 | 1.0 | 17.0 | 3.0 | 2.0 | 2.0 | 4.0 | 4.0 | 15.0 | 76.0 | 32.0 |
| 20 | 4.0 | 9.0 | 13.0 | 2.0 | 16.0 | 5.0 | 10.0 | 9.0 | 19.0 | 19.0 | 58.0 | 11.0 | 17.0 | 8.0 | 28.0 | 17.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 0.482 | 0.211 | -0.757 | -0.903 | 0.726 | -0.516 | -1.899 | -0.322 | 0.262 | -0.516 | -0.414 | -0.414 | 0.558 | 0.211 | -0.02 | 0.441 |
| 02 | 1.148 | -0.629 | 0.726 | -0.02 | 1.063 | -0.903 | -1.899 | -1.544 | -0.088 | -0.903 | -0.903 | -0.629 | 0.482 | -0.629 | -0.322 | -0.757 |
| 03 | 0.726 | 0.043 | 1.148 | 0.842 | 0.158 | -1.544 | -1.544 | -1.282 | -0.02 | -0.322 | 0.102 | -0.629 | -0.903 | -0.629 | -0.088 | -1.075 |
| 04 | 0.896 | -1.544 | 0.482 | -1.075 | 0.558 | -3.818 | -1.282 | -0.757 | 0.921 | -0.238 | 0.31 | -0.322 | 0.356 | -2.455 | 0.558 | -1.544 |
| 05 | 1.018 | -0.414 | 0.869 | 0.842 | -0.516 | -2.455 | -3.818 | -0.903 | 0.211 | -0.02 | -0.903 | 0.869 | -0.903 | -1.899 | -0.322 | -0.757 |
| 06 | -0.629 | -3.818 | 1.382 | -0.757 | -0.629 | -2.455 | -0.757 | -0.238 | -0.757 | -3.818 | 1.063 | -1.899 | -0.516 | -1.544 | 1.429 | -0.516 |
| 07 | -0.238 | -2.455 | 0.211 | -3.818 | -1.899 | -1.899 | -3.818 | -3.818 | 0.971 | -1.544 | 2.135 | -1.544 | -1.075 | -1.544 | 0.158 | -1.282 |
| 08 | -1.282 | 0.756 | -1.544 | 0.262 | -3.818 | -0.903 | -3.818 | -1.544 | -0.414 | 1.827 | -1.075 | 1.298 | -3.818 | -1.282 | -3.818 | -1.544 |
| 09 | -0.516 | -3.818 | -1.075 | -3.818 | 2.164 | -3.818 | -2.455 | -1.544 | -0.629 | -3.818 | -3.818 | -3.818 | 1.244 | -3.818 | 0.558 | -1.899 |
| 10 | -0.903 | -1.899 | 2.534 | -1.899 | -3.818 | -3.818 | -3.818 | -3.818 | -1.899 | -3.818 | 0.694 | -3.818 | -3.818 | -3.818 | -1.282 | -2.455 |
| 11 | -2.455 | -0.757 | -3.818 | -3.818 | -2.455 | -3.818 | -2.455 | -3.818 | -0.903 | 2.006 | -0.16 | 1.827 | -3.818 | -1.899 | -2.455 | -3.818 |
| 12 | -1.282 | -3.818 | -1.282 | -3.818 | 2.048 | -2.455 | -2.455 | -1.899 | -0.757 | -2.455 | -1.075 | -1.899 | 1.459 | -1.544 | 0.399 | -1.282 |
| 13 | 0.842 | 1.127 | 1.817 | 0.211 | -1.544 | -2.455 | -3.818 | -2.455 | -1.899 | -0.629 | 0.158 | -1.075 | -3.818 | -1.899 | -1.075 | -2.455 |
| 14 | -1.899 | 0.814 | -1.899 | -1.899 | -3.818 | 1.332 | -3.818 | -3.818 | -0.757 | 1.944 | -3.818 | -1.544 | -3.818 | 0.482 | -3.818 | -2.455 |
| 15 | -1.544 | -3.818 | -1.544 | -1.544 | 2.142 | 0.102 | -1.899 | 1.584 | -3.818 | -2.455 | -2.455 | -3.818 | -1.075 | -2.455 | -3.818 | -3.818 |
| 16 | -0.238 | 1.366 | 0.694 | 0.869 | -0.757 | -1.544 | -1.544 | -0.903 | -2.455 | -3.818 | -1.899 | -1.544 | -1.544 | -0.238 | 0.043 | 1.127 |
| 17 | -1.282 | -0.088 | -1.075 | -3.818 | -0.757 | 1.413 | -3.818 | -1.075 | -2.455 | 1.063 | -1.282 | -1.899 | -1.544 | 1.684 | -0.757 | -3.818 |
| 18 | -0.903 | -1.544 | -1.899 | -1.282 | 1.188 | 0.726 | -1.075 | 2.031 | -0.629 | -2.455 | -1.544 | -1.282 | -1.282 | -1.899 | -2.455 | -3.818 |
| 19 | -0.629 | 0.043 | 0.594 | 0.102 | -0.16 | -0.757 | -2.455 | 0.102 | -1.544 | -1.899 | -1.899 | -1.282 | -1.282 | -0.02 | 1.584 | 0.726 |
| 20 | -1.282 | -0.516 | -0.16 | -1.899 | 0.043 | -1.075 | -0.414 | -0.516 | 0.211 | 0.211 | 1.315 | -0.322 | 0.102 | -0.629 | 0.594 | 0.102 |