Model info
| Transcription factor | REST (GeneCards) | ||||||||
| Model | REST_HUMAN.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 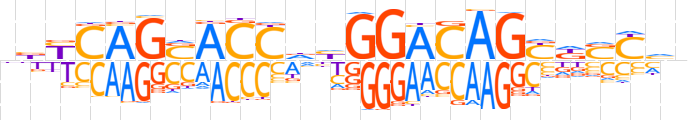 | ||||||||
| LOGO (reverse complement) | 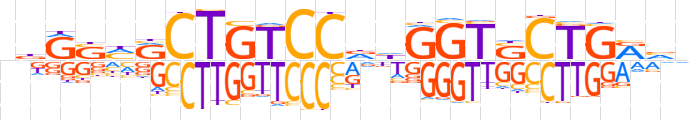 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 24 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | nbYCAGSACCdhGGACAGYdSYhn | ||||||||
| Best auROC (human) | 0.999 | ||||||||
| Best auROC (mouse) | 0.996 | ||||||||
| Peak sets in benchmark (human) | 147 | ||||||||
| Peak sets in benchmark (mouse) | 33 | ||||||||
| Aligned words | 319 | ||||||||
| TF family | Factors with multiple dispersed zinc fingers {2.3.4} | ||||||||
| TF subfamily | unclassified {2.3.4.0} | ||||||||
| HGNC | HGNC:9966 | ||||||||
| EntrezGene | GeneID:5978 (SSTAR profile) | ||||||||
| UniProt ID | REST_HUMAN | ||||||||
| UniProt AC | Q13127 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | REST expression | ||||||||
| ReMap ChIP-seq dataset list | REST datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 6.0 | 21.0 | 14.0 | 26.0 | 17.0 | 8.0 | 20.0 | 42.0 | 9.0 | 10.0 | 39.0 | 38.0 | 1.0 | 7.0 | 7.0 | 54.0 |
| 02 | 0.0 | 12.0 | 8.0 | 13.0 | 4.0 | 19.0 | 3.0 | 20.0 | 8.0 | 16.0 | 12.0 | 44.0 | 0.0 | 13.0 | 9.0 | 138.0 |
| 03 | 0.0 | 10.0 | 2.0 | 0.0 | 4.0 | 42.0 | 7.0 | 7.0 | 2.0 | 21.0 | 5.0 | 4.0 | 5.0 | 205.0 | 4.0 | 1.0 |
| 04 | 3.0 | 2.0 | 5.0 | 1.0 | 264.0 | 8.0 | 4.0 | 2.0 | 7.0 | 7.0 | 1.0 | 3.0 | 3.0 | 2.0 | 4.0 | 3.0 |
| 05 | 2.0 | 1.0 | 274.0 | 0.0 | 8.0 | 6.0 | 3.0 | 2.0 | 0.0 | 4.0 | 10.0 | 0.0 | 0.0 | 3.0 | 6.0 | 0.0 |
| 06 | 3.0 | 2.0 | 5.0 | 0.0 | 6.0 | 3.0 | 2.0 | 3.0 | 17.0 | 211.0 | 45.0 | 20.0 | 0.0 | 0.0 | 2.0 | 0.0 |
| 07 | 13.0 | 3.0 | 10.0 | 0.0 | 207.0 | 1.0 | 3.0 | 5.0 | 42.0 | 5.0 | 5.0 | 2.0 | 19.0 | 0.0 | 2.0 | 2.0 |
| 08 | 0.0 | 276.0 | 4.0 | 1.0 | 0.0 | 0.0 | 3.0 | 6.0 | 1.0 | 5.0 | 7.0 | 7.0 | 0.0 | 1.0 | 6.0 | 2.0 |
| 09 | 1.0 | 0.0 | 0.0 | 0.0 | 4.0 | 263.0 | 2.0 | 13.0 | 6.0 | 6.0 | 6.0 | 2.0 | 0.0 | 6.0 | 8.0 | 2.0 |
| 10 | 4.0 | 1.0 | 6.0 | 0.0 | 158.0 | 23.0 | 45.0 | 49.0 | 0.0 | 10.0 | 4.0 | 2.0 | 1.0 | 4.0 | 9.0 | 3.0 |
| 11 | 28.0 | 55.0 | 11.0 | 69.0 | 1.0 | 4.0 | 0.0 | 33.0 | 9.0 | 32.0 | 5.0 | 18.0 | 5.0 | 16.0 | 4.0 | 29.0 |
| 12 | 3.0 | 1.0 | 39.0 | 0.0 | 4.0 | 2.0 | 97.0 | 4.0 | 2.0 | 0.0 | 18.0 | 0.0 | 1.0 | 0.0 | 146.0 | 2.0 |
| 13 | 0.0 | 0.0 | 9.0 | 1.0 | 0.0 | 1.0 | 1.0 | 1.0 | 5.0 | 0.0 | 293.0 | 2.0 | 1.0 | 0.0 | 4.0 | 1.0 |
| 14 | 2.0 | 0.0 | 3.0 | 1.0 | 1.0 | 0.0 | 0.0 | 0.0 | 272.0 | 19.0 | 4.0 | 12.0 | 0.0 | 0.0 | 1.0 | 4.0 |
| 15 | 0.0 | 236.0 | 34.0 | 5.0 | 1.0 | 17.0 | 1.0 | 0.0 | 0.0 | 5.0 | 3.0 | 0.0 | 0.0 | 12.0 | 4.0 | 1.0 |
| 16 | 0.0 | 0.0 | 1.0 | 0.0 | 265.0 | 3.0 | 2.0 | 0.0 | 34.0 | 4.0 | 3.0 | 1.0 | 5.0 | 0.0 | 1.0 | 0.0 |
| 17 | 6.0 | 3.0 | 294.0 | 1.0 | 0.0 | 0.0 | 5.0 | 2.0 | 2.0 | 1.0 | 2.0 | 2.0 | 0.0 | 0.0 | 1.0 | 0.0 |
| 18 | 0.0 | 3.0 | 5.0 | 0.0 | 0.0 | 1.0 | 1.0 | 2.0 | 25.0 | 221.0 | 22.0 | 34.0 | 0.0 | 1.0 | 4.0 | 0.0 |
| 19 | 1.0 | 2.0 | 19.0 | 3.0 | 56.0 | 2.0 | 84.0 | 84.0 | 7.0 | 0.0 | 21.0 | 4.0 | 5.0 | 0.0 | 21.0 | 10.0 |
| 20 | 7.0 | 37.0 | 21.0 | 4.0 | 2.0 | 1.0 | 1.0 | 0.0 | 25.0 | 78.0 | 34.0 | 8.0 | 3.0 | 94.0 | 2.0 | 2.0 |
| 21 | 1.0 | 29.0 | 1.0 | 6.0 | 24.0 | 168.0 | 1.0 | 17.0 | 6.0 | 38.0 | 5.0 | 9.0 | 0.0 | 13.0 | 0.0 | 1.0 |
| 22 | 5.0 | 14.0 | 10.0 | 2.0 | 91.0 | 103.0 | 12.0 | 42.0 | 1.0 | 4.0 | 1.0 | 1.0 | 5.0 | 14.0 | 7.0 | 7.0 |
| 23 | 13.0 | 32.0 | 40.0 | 17.0 | 52.0 | 38.0 | 23.0 | 22.0 | 4.0 | 15.0 | 6.0 | 5.0 | 2.0 | 12.0 | 30.0 | 8.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -1.16 | 0.051 | -0.346 | 0.261 | -0.156 | -0.887 | 0.003 | 0.736 | -0.774 | -0.673 | 0.662 | 0.637 | -2.703 | -1.014 | -1.014 | 0.985 |
| 02 | -4.031 | -0.496 | -0.887 | -0.418 | -1.538 | -0.047 | -1.798 | 0.003 | -0.887 | -0.216 | -0.496 | 0.782 | -4.031 | -0.418 | -0.774 | 1.919 |
| 03 | -4.031 | -0.673 | -2.152 | -4.031 | -1.538 | 0.736 | -1.014 | -1.014 | -2.152 | 0.051 | -1.331 | -1.538 | -1.331 | 2.314 | -1.538 | -2.703 |
| 04 | -1.798 | -2.152 | -1.331 | -2.703 | 2.567 | -0.887 | -1.538 | -2.152 | -1.014 | -1.014 | -2.703 | -1.798 | -1.798 | -2.152 | -1.538 | -1.798 |
| 05 | -2.152 | -2.703 | 2.604 | -4.031 | -0.887 | -1.16 | -1.798 | -2.152 | -4.031 | -1.538 | -0.673 | -4.031 | -4.031 | -1.798 | -1.16 | -4.031 |
| 06 | -1.798 | -2.152 | -1.331 | -4.031 | -1.16 | -1.798 | -2.152 | -1.798 | -0.156 | 2.343 | 0.804 | 0.003 | -4.031 | -4.031 | -2.152 | -4.031 |
| 07 | -0.418 | -1.798 | -0.673 | -4.031 | 2.324 | -2.703 | -1.798 | -1.331 | 0.736 | -1.331 | -1.331 | -2.152 | -0.047 | -4.031 | -2.152 | -2.152 |
| 08 | -4.031 | 2.611 | -1.538 | -2.703 | -4.031 | -4.031 | -1.798 | -1.16 | -2.703 | -1.331 | -1.014 | -1.014 | -4.031 | -2.703 | -1.16 | -2.152 |
| 09 | -2.703 | -4.031 | -4.031 | -4.031 | -1.538 | 2.563 | -2.152 | -0.418 | -1.16 | -1.16 | -1.16 | -2.152 | -4.031 | -1.16 | -0.887 | -2.152 |
| 10 | -1.538 | -2.703 | -1.16 | -4.031 | 2.054 | 0.141 | 0.804 | 0.889 | -4.031 | -0.673 | -1.538 | -2.152 | -2.703 | -1.538 | -0.774 | -1.798 |
| 11 | 0.334 | 1.003 | -0.58 | 1.229 | -2.703 | -1.538 | -4.031 | 0.497 | -0.774 | 0.466 | -1.331 | -0.1 | -1.331 | -0.216 | -1.538 | 0.369 |
| 12 | -1.798 | -2.703 | 0.662 | -4.031 | -1.538 | -2.152 | 1.568 | -1.538 | -2.152 | -4.031 | -0.1 | -4.031 | -2.703 | -4.031 | 1.976 | -2.152 |
| 13 | -4.031 | -4.031 | -0.774 | -2.703 | -4.031 | -2.703 | -2.703 | -2.703 | -1.331 | -4.031 | 2.671 | -2.152 | -2.703 | -4.031 | -1.538 | -2.703 |
| 14 | -2.152 | -4.031 | -1.798 | -2.703 | -2.703 | -4.031 | -4.031 | -4.031 | 2.597 | -0.047 | -1.538 | -0.496 | -4.031 | -4.031 | -2.703 | -1.538 |
| 15 | -4.031 | 2.455 | 0.526 | -1.331 | -2.703 | -0.156 | -2.703 | -4.031 | -4.031 | -1.331 | -1.798 | -4.031 | -4.031 | -0.496 | -1.538 | -2.703 |
| 16 | -4.031 | -4.031 | -2.703 | -4.031 | 2.571 | -1.798 | -2.152 | -4.031 | 0.526 | -1.538 | -1.798 | -2.703 | -1.331 | -4.031 | -2.703 | -4.031 |
| 17 | -1.16 | -1.798 | 2.674 | -2.703 | -4.031 | -4.031 | -1.331 | -2.152 | -2.152 | -2.703 | -2.152 | -2.152 | -4.031 | -4.031 | -2.703 | -4.031 |
| 18 | -4.031 | -1.798 | -1.331 | -4.031 | -4.031 | -2.703 | -2.703 | -2.152 | 0.223 | 2.389 | 0.097 | 0.526 | -4.031 | -2.703 | -1.538 | -4.031 |
| 19 | -2.703 | -2.152 | -0.047 | -1.798 | 1.021 | -2.152 | 1.425 | 1.425 | -1.014 | -4.031 | 0.051 | -1.538 | -1.331 | -4.031 | 0.051 | -0.673 |
| 20 | -1.014 | 0.61 | 0.051 | -1.538 | -2.152 | -2.703 | -2.703 | -4.031 | 0.223 | 1.351 | 0.526 | -0.887 | -1.798 | 1.537 | -2.152 | -2.152 |
| 21 | -2.703 | 0.369 | -2.703 | -1.16 | 0.182 | 2.116 | -2.703 | -0.156 | -1.16 | 0.637 | -1.331 | -0.774 | -4.031 | -0.418 | -4.031 | -2.703 |
| 22 | -1.331 | -0.346 | -0.673 | -2.152 | 1.504 | 1.628 | -0.496 | 0.736 | -2.703 | -1.538 | -2.703 | -2.703 | -1.331 | -0.346 | -1.014 | -1.014 |
| 23 | -0.418 | 0.466 | 0.687 | -0.156 | 0.948 | 0.637 | 0.141 | 0.097 | -1.538 | -0.279 | -1.16 | -1.331 | -2.152 | -0.496 | 0.403 | -0.887 |