Model info
| Transcription factor | Rorc | ||||||||
| Model | RORG_MOUSE.H11DI.0.B | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 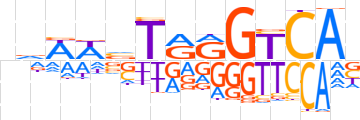 | ||||||||
| LOGO (reverse complement) |  | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 13 | ||||||||
| Quality | B | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | ndWWvTRRGTCAn | ||||||||
| Best auROC (human) | |||||||||
| Best auROC (mouse) | 0.928 | ||||||||
| Peak sets in benchmark (human) | |||||||||
| Peak sets in benchmark (mouse) | 19 | ||||||||
| Aligned words | 504 | ||||||||
| TF family | Thyroid hormone receptor-related factors (NR1) {2.1.2} | ||||||||
| TF subfamily | ROR (NR1F) {2.1.2.6} | ||||||||
| MGI | MGI:104856 | ||||||||
| EntrezGene | GeneID:19885 (SSTAR profile) | ||||||||
| UniProt ID | RORG_MOUSE | ||||||||
| UniProt AC | P51450 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | Rorc expression | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 61.0 | 11.0 | 39.0 | 14.0 | 53.0 | 14.0 | 6.0 | 29.0 | 94.0 | 13.0 | 50.0 | 28.0 | 21.0 | 18.0 | 32.0 | 17.0 |
| 02 | 180.0 | 8.0 | 27.0 | 14.0 | 34.0 | 10.0 | 1.0 | 11.0 | 86.0 | 4.0 | 19.0 | 18.0 | 60.0 | 5.0 | 7.0 | 16.0 |
| 03 | 244.0 | 4.0 | 12.0 | 100.0 | 11.0 | 3.0 | 0.0 | 13.0 | 27.0 | 5.0 | 12.0 | 10.0 | 22.0 | 1.0 | 11.0 | 25.0 |
| 04 | 37.0 | 117.0 | 131.0 | 19.0 | 2.0 | 7.0 | 2.0 | 2.0 | 8.0 | 18.0 | 8.0 | 1.0 | 13.0 | 56.0 | 60.0 | 19.0 |
| 05 | 0.0 | 2.0 | 5.0 | 53.0 | 7.0 | 5.0 | 3.0 | 183.0 | 3.0 | 9.0 | 5.0 | 184.0 | 1.0 | 2.0 | 8.0 | 30.0 |
| 06 | 4.0 | 1.0 | 6.0 | 0.0 | 13.0 | 1.0 | 4.0 | 0.0 | 16.0 | 0.0 | 5.0 | 0.0 | 201.0 | 3.0 | 240.0 | 6.0 |
| 07 | 4.0 | 2.0 | 223.0 | 5.0 | 3.0 | 0.0 | 0.0 | 2.0 | 127.0 | 1.0 | 108.0 | 19.0 | 6.0 | 0.0 | 0.0 | 0.0 |
| 08 | 1.0 | 0.0 | 136.0 | 3.0 | 1.0 | 0.0 | 2.0 | 0.0 | 3.0 | 1.0 | 322.0 | 5.0 | 0.0 | 0.0 | 26.0 | 0.0 |
| 09 | 0.0 | 0.0 | 3.0 | 2.0 | 0.0 | 0.0 | 0.0 | 1.0 | 15.0 | 42.0 | 36.0 | 393.0 | 0.0 | 0.0 | 0.0 | 8.0 |
| 10 | 0.0 | 15.0 | 0.0 | 0.0 | 0.0 | 41.0 | 0.0 | 1.0 | 0.0 | 38.0 | 1.0 | 0.0 | 2.0 | 380.0 | 4.0 | 18.0 |
| 11 | 2.0 | 0.0 | 0.0 | 0.0 | 458.0 | 1.0 | 12.0 | 3.0 | 5.0 | 0.0 | 0.0 | 0.0 | 15.0 | 0.0 | 0.0 | 4.0 |
| 12 | 72.0 | 141.0 | 152.0 | 115.0 | 0.0 | 0.0 | 1.0 | 0.0 | 1.0 | 1.0 | 3.0 | 7.0 | 1.0 | 4.0 | 2.0 | 0.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 0.663 | -1.022 | 0.219 | -0.788 | 0.523 | -0.788 | -1.6 | -0.074 | 1.093 | -0.86 | 0.465 | -0.108 | -0.392 | -0.543 | 0.023 | -0.599 |
| 02 | 1.741 | -1.328 | -0.144 | -0.788 | 0.083 | -1.114 | -3.126 | -1.022 | 1.004 | -1.975 | -0.49 | -0.543 | 0.646 | -1.77 | -1.454 | -0.658 |
| 03 | 2.044 | -1.975 | -0.938 | 1.155 | -1.022 | -2.234 | -4.4 | -0.86 | -0.144 | -1.77 | -0.938 | -1.114 | -0.346 | -3.126 | -1.022 | -0.22 |
| 04 | 0.167 | 1.311 | 1.424 | -0.49 | -2.584 | -1.454 | -2.584 | -2.584 | -1.328 | -0.543 | -1.328 | -3.126 | -0.86 | 0.578 | 0.646 | -0.49 |
| 05 | -4.4 | -2.584 | -1.77 | 0.523 | -1.454 | -1.77 | -2.234 | 1.757 | -2.234 | -1.215 | -1.77 | 1.763 | -3.126 | -2.584 | -1.328 | -0.04 |
| 06 | -1.975 | -3.126 | -1.6 | -4.4 | -0.86 | -3.126 | -1.975 | -4.4 | -0.658 | -4.4 | -1.77 | -4.4 | 1.851 | -2.234 | 2.028 | -1.6 |
| 07 | -1.975 | -2.584 | 1.955 | -1.77 | -2.234 | -4.4 | -4.4 | -2.584 | 1.393 | -3.126 | 1.231 | -0.49 | -1.6 | -4.4 | -4.4 | -4.4 |
| 08 | -3.126 | -4.4 | 1.461 | -2.234 | -3.126 | -4.4 | -2.584 | -4.4 | -2.234 | -3.126 | 2.321 | -1.77 | -4.4 | -4.4 | -0.181 | -4.4 |
| 09 | -4.4 | -4.4 | -2.234 | -2.584 | -4.4 | -4.4 | -4.4 | -3.126 | -0.721 | 0.293 | 0.14 | 2.52 | -4.4 | -4.4 | -4.4 | -1.328 |
| 10 | -4.4 | -0.721 | -4.4 | -4.4 | -4.4 | 0.269 | -4.4 | -3.126 | -4.4 | 0.193 | -3.126 | -4.4 | -2.584 | 2.487 | -1.975 | -0.543 |
| 11 | -2.584 | -4.4 | -4.4 | -4.4 | 2.673 | -3.126 | -0.938 | -2.234 | -1.77 | -4.4 | -4.4 | -4.4 | -0.721 | -4.4 | -4.4 | -1.975 |
| 12 | 0.828 | 1.497 | 1.572 | 1.294 | -4.4 | -4.4 | -3.126 | -4.4 | -3.126 | -3.126 | -2.234 | -1.454 | -3.126 | -1.975 | -2.584 | -4.4 |