Model info
| Transcription factor | Spib | ||||||||
| Model | SPIB_MOUSE.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 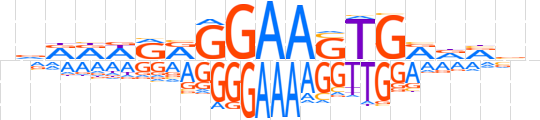 | ||||||||
| LOGO (reverse complement) |  | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 19 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | nvRRWRRGGAASTGRdRvn | ||||||||
| Best auROC (human) | 0.999 | ||||||||
| Best auROC (mouse) | 0.983 | ||||||||
| Peak sets in benchmark (human) | 12 | ||||||||
| Peak sets in benchmark (mouse) | 3 | ||||||||
| Aligned words | 500 | ||||||||
| TF family | Ets-related factors {3.5.2} | ||||||||
| TF subfamily | Spi-like factors {3.5.2.5} | ||||||||
| MGI | MGI:892986 | ||||||||
| EntrezGene | GeneID:272382 (SSTAR profile) | ||||||||
| UniProt ID | SPIB_MOUSE | ||||||||
| UniProt AC | O35906 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | Spib expression | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 80.0 | 15.0 | 88.0 | 8.0 | 33.0 | 19.0 | 21.0 | 18.0 | 60.0 | 23.0 | 37.0 | 8.0 | 22.0 | 18.0 | 35.0 | 13.0 |
| 02 | 139.0 | 6.0 | 32.0 | 18.0 | 41.0 | 8.0 | 7.0 | 19.0 | 129.0 | 8.0 | 25.0 | 19.0 | 17.0 | 5.0 | 11.0 | 14.0 |
| 03 | 257.0 | 12.0 | 42.0 | 15.0 | 16.0 | 3.0 | 3.0 | 5.0 | 38.0 | 13.0 | 17.0 | 7.0 | 19.0 | 7.0 | 18.0 | 26.0 |
| 04 | 232.0 | 8.0 | 37.0 | 53.0 | 27.0 | 1.0 | 1.0 | 6.0 | 43.0 | 4.0 | 27.0 | 6.0 | 14.0 | 0.0 | 6.0 | 33.0 |
| 05 | 51.0 | 18.0 | 238.0 | 9.0 | 9.0 | 3.0 | 1.0 | 0.0 | 19.0 | 9.0 | 42.0 | 1.0 | 18.0 | 2.0 | 68.0 | 10.0 |
| 06 | 42.0 | 7.0 | 47.0 | 1.0 | 26.0 | 1.0 | 5.0 | 0.0 | 233.0 | 45.0 | 67.0 | 4.0 | 9.0 | 0.0 | 11.0 | 0.0 |
| 07 | 11.0 | 1.0 | 297.0 | 1.0 | 36.0 | 0.0 | 17.0 | 0.0 | 27.0 | 0.0 | 103.0 | 0.0 | 0.0 | 1.0 | 4.0 | 0.0 |
| 08 | 1.0 | 0.0 | 72.0 | 1.0 | 0.0 | 0.0 | 2.0 | 0.0 | 0.0 | 5.0 | 416.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 |
| 09 | 2.0 | 0.0 | 0.0 | 0.0 | 4.0 | 0.0 | 0.0 | 1.0 | 488.0 | 1.0 | 1.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 |
| 10 | 485.0 | 0.0 | 2.0 | 8.0 | 0.0 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 |
| 11 | 36.0 | 83.0 | 366.0 | 2.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 5.0 | 0.0 | 3.0 | 0.0 |
| 12 | 2.0 | 3.0 | 0.0 | 36.0 | 10.0 | 2.0 | 0.0 | 71.0 | 22.0 | 8.0 | 0.0 | 342.0 | 0.0 | 1.0 | 0.0 | 1.0 |
| 13 | 2.0 | 2.0 | 30.0 | 0.0 | 1.0 | 3.0 | 9.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 4.0 | 19.0 | 425.0 | 2.0 |
| 14 | 3.0 | 0.0 | 4.0 | 0.0 | 8.0 | 3.0 | 7.0 | 6.0 | 318.0 | 48.0 | 77.0 | 21.0 | 1.0 | 0.0 | 2.0 | 0.0 |
| 15 | 234.0 | 12.0 | 68.0 | 16.0 | 17.0 | 8.0 | 5.0 | 21.0 | 24.0 | 14.0 | 37.0 | 15.0 | 4.0 | 7.0 | 10.0 | 6.0 |
| 16 | 227.0 | 11.0 | 27.0 | 14.0 | 11.0 | 13.0 | 5.0 | 12.0 | 63.0 | 17.0 | 26.0 | 14.0 | 13.0 | 19.0 | 16.0 | 10.0 |
| 17 | 49.0 | 135.0 | 114.0 | 16.0 | 21.0 | 19.0 | 5.0 | 15.0 | 17.0 | 19.0 | 17.0 | 21.0 | 13.0 | 16.0 | 13.0 | 8.0 |
| 18 | 24.0 | 22.0 | 39.0 | 15.0 | 32.0 | 71.0 | 13.0 | 73.0 | 36.0 | 32.0 | 26.0 | 55.0 | 8.0 | 20.0 | 21.0 | 11.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 0.936 | -0.717 | 1.031 | -1.324 | 0.058 | -0.486 | -0.388 | -0.539 | 0.65 | -0.298 | 0.171 | -1.324 | -0.342 | -0.539 | 0.116 | -0.856 |
| 02 | 1.487 | -1.596 | 0.027 | -0.539 | 0.273 | -1.324 | -1.451 | -0.486 | 1.412 | -1.324 | -0.216 | -0.486 | -0.595 | -1.766 | -1.018 | -0.784 |
| 03 | 2.1 | -0.934 | 0.296 | -0.717 | -0.654 | -2.23 | -2.23 | -1.766 | 0.197 | -0.856 | -0.595 | -1.451 | -0.486 | -1.451 | -0.539 | -0.177 |
| 04 | 1.998 | -1.324 | 0.171 | 0.527 | -0.14 | -3.122 | -3.122 | -1.596 | 0.32 | -1.971 | -0.14 | -1.596 | -0.784 | -4.397 | -1.596 | 0.058 |
| 05 | 0.489 | -0.539 | 2.023 | -1.211 | -1.211 | -2.23 | -3.122 | -4.397 | -0.486 | -1.211 | 0.296 | -3.122 | -0.539 | -2.58 | 0.775 | -1.11 |
| 06 | 0.296 | -1.451 | 0.408 | -3.122 | -0.177 | -3.122 | -1.766 | -4.397 | 2.002 | 0.365 | 0.76 | -1.971 | -1.211 | -4.397 | -1.018 | -4.397 |
| 07 | -1.018 | -3.122 | 2.245 | -3.122 | 0.144 | -4.397 | -0.595 | -4.397 | -0.14 | -4.397 | 1.188 | -4.397 | -4.397 | -3.122 | -1.971 | -4.397 |
| 08 | -3.122 | -4.397 | 0.832 | -3.122 | -4.397 | -4.397 | -2.58 | -4.397 | -4.397 | -1.766 | 2.581 | -4.397 | -3.122 | -4.397 | -4.397 | -4.397 |
| 09 | -2.58 | -4.397 | -4.397 | -4.397 | -1.971 | -4.397 | -4.397 | -3.122 | 2.741 | -3.122 | -3.122 | -4.397 | -3.122 | -4.397 | -4.397 | -4.397 |
| 10 | 2.735 | -4.397 | -2.58 | -1.324 | -4.397 | -4.397 | -3.122 | -4.397 | -3.122 | -4.397 | -4.397 | -4.397 | -3.122 | -4.397 | -4.397 | -4.397 |
| 11 | 0.144 | 0.973 | 2.453 | -2.58 | -4.397 | -4.397 | -4.397 | -4.397 | -4.397 | -4.397 | -2.23 | -4.397 | -1.766 | -4.397 | -2.23 | -4.397 |
| 12 | -2.58 | -2.23 | -4.397 | 0.144 | -1.11 | -2.58 | -4.397 | 0.818 | -0.342 | -1.324 | -4.397 | 2.386 | -4.397 | -3.122 | -4.397 | -3.122 |
| 13 | -2.58 | -2.58 | -0.036 | -4.397 | -3.122 | -2.23 | -1.211 | -3.122 | -4.397 | -4.397 | -4.397 | -4.397 | -1.971 | -0.486 | 2.603 | -2.58 |
| 14 | -2.23 | -4.397 | -1.971 | -4.397 | -1.324 | -2.23 | -1.451 | -1.596 | 2.313 | 0.429 | 0.898 | -0.388 | -3.122 | -4.397 | -2.58 | -4.397 |
| 15 | 2.007 | -0.934 | 0.775 | -0.654 | -0.595 | -1.324 | -1.766 | -0.388 | -0.256 | -0.784 | 0.171 | -0.717 | -1.971 | -1.451 | -1.11 | -1.596 |
| 16 | 1.976 | -1.018 | -0.14 | -0.784 | -1.018 | -0.856 | -1.766 | -0.934 | 0.699 | -0.595 | -0.177 | -0.784 | -0.856 | -0.486 | -0.654 | -1.11 |
| 17 | 0.449 | 1.458 | 1.289 | -0.654 | -0.388 | -0.486 | -1.766 | -0.717 | -0.595 | -0.486 | -0.595 | -0.388 | -0.856 | -0.654 | -0.856 | -1.324 |
| 18 | -0.256 | -0.342 | 0.223 | -0.717 | 0.027 | 0.818 | -0.856 | 0.845 | 0.144 | 0.027 | -0.177 | 0.564 | -1.324 | -0.435 | -0.388 | -1.018 |