Model info
| Transcription factor | Zbtb17 | ||||||||
| Model | ZBT17_MOUSE.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 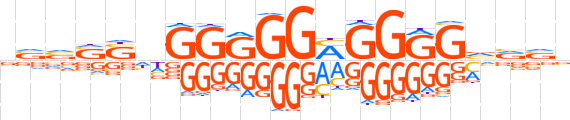 | ||||||||
| LOGO (reverse complement) | 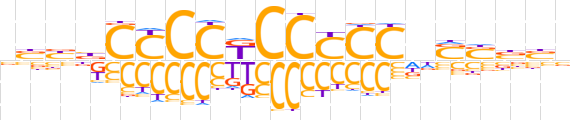 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 20 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | vvvSSnGGRGGMGGGGvvvv | ||||||||
| Best auROC (human) | 0.947 | ||||||||
| Best auROC (mouse) | 0.942 | ||||||||
| Peak sets in benchmark (human) | 21 | ||||||||
| Peak sets in benchmark (mouse) | 25 | ||||||||
| Aligned words | 498 | ||||||||
| TF family | Factors with multiple dispersed zinc fingers {2.3.4} | ||||||||
| TF subfamily | unclassified {2.3.4.0} | ||||||||
| MGI | MGI:107410 | ||||||||
| EntrezGene | GeneID:22642 (SSTAR profile) | ||||||||
| UniProt ID | ZBT17_MOUSE | ||||||||
| UniProt AC | Q60821 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | Zbtb17 expression | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 11.0 | 10.0 | 36.0 | 2.0 | 33.0 | 37.0 | 73.0 | 12.0 | 28.0 | 44.0 | 143.0 | 18.0 | 7.0 | 12.0 | 23.0 | 6.0 |
| 02 | 17.0 | 10.0 | 50.0 | 2.0 | 22.0 | 21.0 | 51.0 | 9.0 | 65.0 | 107.0 | 99.0 | 4.0 | 3.0 | 6.0 | 25.0 | 4.0 |
| 03 | 18.0 | 4.0 | 81.0 | 4.0 | 14.0 | 24.0 | 98.0 | 8.0 | 36.0 | 41.0 | 127.0 | 21.0 | 1.0 | 4.0 | 11.0 | 3.0 |
| 04 | 7.0 | 5.0 | 48.0 | 9.0 | 8.0 | 6.0 | 47.0 | 12.0 | 24.0 | 57.0 | 212.0 | 24.0 | 3.0 | 3.0 | 27.0 | 3.0 |
| 05 | 8.0 | 5.0 | 16.0 | 13.0 | 3.0 | 13.0 | 22.0 | 33.0 | 117.0 | 80.0 | 25.0 | 112.0 | 6.0 | 6.0 | 22.0 | 14.0 |
| 06 | 20.0 | 2.0 | 108.0 | 4.0 | 6.0 | 1.0 | 88.0 | 9.0 | 10.0 | 4.0 | 71.0 | 0.0 | 9.0 | 4.0 | 157.0 | 2.0 |
| 07 | 7.0 | 6.0 | 31.0 | 1.0 | 1.0 | 3.0 | 6.0 | 1.0 | 24.0 | 6.0 | 378.0 | 16.0 | 1.0 | 1.0 | 12.0 | 1.0 |
| 08 | 6.0 | 3.0 | 21.0 | 3.0 | 6.0 | 0.0 | 10.0 | 0.0 | 81.0 | 8.0 | 327.0 | 11.0 | 0.0 | 2.0 | 17.0 | 0.0 |
| 09 | 6.0 | 2.0 | 85.0 | 0.0 | 2.0 | 1.0 | 9.0 | 1.0 | 7.0 | 4.0 | 357.0 | 7.0 | 2.0 | 0.0 | 12.0 | 0.0 |
| 10 | 2.0 | 0.0 | 15.0 | 0.0 | 0.0 | 1.0 | 6.0 | 0.0 | 5.0 | 2.0 | 454.0 | 2.0 | 0.0 | 1.0 | 7.0 | 0.0 |
| 11 | 6.0 | 1.0 | 0.0 | 0.0 | 1.0 | 3.0 | 0.0 | 0.0 | 293.0 | 146.0 | 3.0 | 40.0 | 1.0 | 0.0 | 0.0 | 1.0 |
| 12 | 28.0 | 2.0 | 263.0 | 8.0 | 12.0 | 1.0 | 127.0 | 10.0 | 0.0 | 0.0 | 2.0 | 1.0 | 0.0 | 0.0 | 40.0 | 1.0 |
| 13 | 4.0 | 0.0 | 35.0 | 1.0 | 1.0 | 0.0 | 2.0 | 0.0 | 1.0 | 5.0 | 418.0 | 8.0 | 1.0 | 0.0 | 19.0 | 0.0 |
| 14 | 4.0 | 0.0 | 0.0 | 3.0 | 2.0 | 0.0 | 2.0 | 1.0 | 70.0 | 5.0 | 378.0 | 21.0 | 0.0 | 0.0 | 8.0 | 1.0 |
| 15 | 2.0 | 4.0 | 66.0 | 4.0 | 0.0 | 2.0 | 2.0 | 1.0 | 20.0 | 21.0 | 345.0 | 2.0 | 1.0 | 2.0 | 22.0 | 1.0 |
| 16 | 9.0 | 8.0 | 6.0 | 0.0 | 4.0 | 8.0 | 7.0 | 10.0 | 158.0 | 197.0 | 50.0 | 30.0 | 1.0 | 2.0 | 4.0 | 1.0 |
| 17 | 33.0 | 19.0 | 115.0 | 5.0 | 44.0 | 38.0 | 111.0 | 22.0 | 17.0 | 19.0 | 23.0 | 8.0 | 6.0 | 8.0 | 26.0 | 1.0 |
| 18 | 19.0 | 15.0 | 60.0 | 6.0 | 13.0 | 5.0 | 50.0 | 16.0 | 46.0 | 67.0 | 146.0 | 16.0 | 7.0 | 8.0 | 18.0 | 3.0 |
| 19 | 15.0 | 20.0 | 44.0 | 6.0 | 9.0 | 23.0 | 45.0 | 18.0 | 52.0 | 83.0 | 111.0 | 28.0 | 7.0 | 12.0 | 22.0 | 0.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -1.012 | -1.104 | 0.15 | -2.574 | 0.064 | 0.177 | 0.851 | -0.928 | -0.098 | 0.349 | 1.521 | -0.533 | -1.445 | -0.928 | -0.292 | -1.59 |
| 02 | -0.589 | -1.104 | 0.475 | -2.574 | -0.336 | -0.382 | 0.495 | -1.205 | 0.736 | 1.232 | 1.155 | -1.966 | -2.224 | -1.59 | -0.21 | -1.966 |
| 03 | -0.533 | -1.966 | 0.955 | -1.966 | -0.778 | -0.25 | 1.144 | -1.318 | 0.15 | 0.279 | 1.403 | -0.382 | -3.117 | -1.966 | -1.012 | -2.224 |
| 04 | -1.445 | -1.76 | 0.435 | -1.205 | -1.318 | -1.59 | 0.414 | -0.928 | -0.25 | 0.605 | 1.914 | -0.25 | -2.224 | -2.224 | -0.134 | -2.224 |
| 05 | -1.318 | -1.76 | -0.648 | -0.85 | -2.224 | -0.85 | -0.336 | 0.064 | 1.321 | 0.942 | -0.21 | 1.278 | -1.59 | -1.59 | -0.336 | -0.778 |
| 06 | -0.429 | -2.574 | 1.241 | -1.966 | -1.59 | -3.117 | 1.037 | -1.205 | -1.104 | -1.966 | 0.824 | -4.392 | -1.205 | -1.966 | 1.614 | -2.574 |
| 07 | -1.445 | -1.59 | 0.002 | -3.117 | -3.117 | -2.224 | -1.59 | -3.117 | -0.25 | -1.59 | 2.491 | -0.648 | -3.117 | -3.117 | -0.928 | -3.117 |
| 08 | -1.59 | -2.224 | -0.382 | -2.224 | -1.59 | -4.392 | -1.104 | -4.392 | 0.955 | -1.318 | 2.347 | -1.012 | -4.392 | -2.574 | -0.589 | -4.392 |
| 09 | -1.59 | -2.574 | 1.003 | -4.392 | -2.574 | -3.117 | -1.205 | -3.117 | -1.445 | -1.966 | 2.434 | -1.445 | -2.574 | -4.392 | -0.928 | -4.392 |
| 10 | -2.574 | -4.392 | -0.711 | -4.392 | -4.392 | -3.117 | -1.59 | -4.392 | -1.76 | -2.574 | 2.675 | -2.574 | -4.392 | -3.117 | -1.445 | -4.392 |
| 11 | -1.59 | -3.117 | -4.392 | -4.392 | -3.117 | -2.224 | -4.392 | -4.392 | 2.237 | 1.542 | -2.224 | 0.254 | -3.117 | -4.392 | -4.392 | -3.117 |
| 12 | -0.098 | -2.574 | 2.129 | -1.318 | -0.928 | -3.117 | 1.403 | -1.104 | -4.392 | -4.392 | -2.574 | -3.117 | -4.392 | -4.392 | 0.254 | -3.117 |
| 13 | -1.966 | -4.392 | 0.122 | -3.117 | -3.117 | -4.392 | -2.574 | -4.392 | -3.117 | -1.76 | 2.592 | -1.318 | -3.117 | -4.392 | -0.48 | -4.392 |
| 14 | -1.966 | -4.392 | -4.392 | -2.224 | -2.574 | -4.392 | -2.574 | -3.117 | 0.81 | -1.76 | 2.491 | -0.382 | -4.392 | -4.392 | -1.318 | -3.117 |
| 15 | -2.574 | -1.966 | 0.751 | -1.966 | -4.392 | -2.574 | -2.574 | -3.117 | -0.429 | -0.382 | 2.4 | -2.574 | -3.117 | -2.574 | -0.336 | -3.117 |
| 16 | -1.205 | -1.318 | -1.59 | -4.392 | -1.966 | -1.318 | -1.445 | -1.104 | 1.621 | 1.841 | 0.475 | -0.03 | -3.117 | -2.574 | -1.966 | -3.117 |
| 17 | 0.064 | -0.48 | 1.304 | -1.76 | 0.349 | 0.203 | 1.269 | -0.336 | -0.589 | -0.48 | -0.292 | -1.318 | -1.59 | -1.318 | -0.172 | -3.117 |
| 18 | -0.48 | -0.711 | 0.656 | -1.59 | -0.85 | -1.76 | 0.475 | -0.648 | 0.393 | 0.766 | 1.542 | -0.648 | -1.445 | -1.318 | -0.533 | -2.224 |
| 19 | -0.711 | -0.429 | 0.349 | -1.59 | -1.205 | -0.292 | 0.371 | -0.533 | 0.514 | 0.979 | 1.269 | -0.098 | -1.445 | -0.928 | -0.336 | -4.392 |