Model info
| Transcription factor | ZNF140 (GeneCards) | ||||||||
| Model | ZN140_HUMAN.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 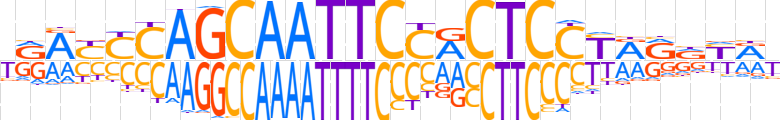 | ||||||||
| LOGO (reverse complement) | 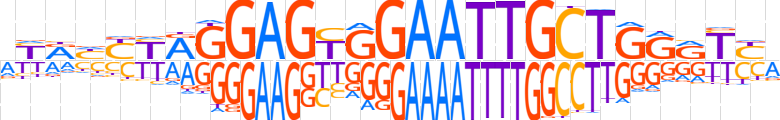 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 27 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | YRAYYCAGCAATTCCACTCCYRRRYRY | ||||||||
| Best auROC (human) | 0.98 | ||||||||
| Best auROC (mouse) | |||||||||
| Peak sets in benchmark (human) | 4 | ||||||||
| Peak sets in benchmark (mouse) | |||||||||
| Aligned words | 249 | ||||||||
| TF family | More than 3 adjacent zinc finger factors {2.3.3} | ||||||||
| TF subfamily | ZNF302-like factors {2.3.3.44} | ||||||||
| HGNC | HGNC:12925 | ||||||||
| EntrezGene | GeneID:7699 (SSTAR profile) | ||||||||
| UniProt ID | ZN140_HUMAN | ||||||||
| UniProt AC | P52738 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | ZNF140 expression | ||||||||
| ReMap ChIP-seq dataset list | ZNF140 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 2.0 | 5.0 | 3.0 | 8.0 | 21.0 | 4.0 | 8.0 | 2.0 | 7.0 | 0.0 | 14.0 | 2.0 | 22.0 | 6.0 | 142.0 | 3.0 |
| 02 | 33.0 | 6.0 | 10.0 | 3.0 | 8.0 | 5.0 | 0.0 | 2.0 | 154.0 | 4.0 | 5.0 | 4.0 | 9.0 | 1.0 | 5.0 | 0.0 |
| 03 | 5.0 | 133.0 | 12.0 | 54.0 | 2.0 | 7.0 | 0.0 | 7.0 | 2.0 | 13.0 | 2.0 | 3.0 | 2.0 | 6.0 | 0.0 | 1.0 |
| 04 | 0.0 | 7.0 | 1.0 | 3.0 | 5.0 | 128.0 | 1.0 | 25.0 | 0.0 | 9.0 | 2.0 | 3.0 | 5.0 | 47.0 | 4.0 | 9.0 |
| 05 | 0.0 | 6.0 | 1.0 | 3.0 | 3.0 | 170.0 | 0.0 | 18.0 | 0.0 | 6.0 | 1.0 | 1.0 | 0.0 | 29.0 | 3.0 | 8.0 |
| 06 | 3.0 | 0.0 | 0.0 | 0.0 | 204.0 | 1.0 | 1.0 | 5.0 | 4.0 | 0.0 | 1.0 | 0.0 | 27.0 | 1.0 | 1.0 | 1.0 |
| 07 | 9.0 | 0.0 | 229.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 1.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 | 6.0 | 0.0 |
| 08 | 0.0 | 10.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 239.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 09 | 0.0 | 0.0 | 0.0 | 0.0 | 247.0 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 10 | 244.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 |
| 11 | 0.0 | 0.0 | 0.0 | 246.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 12 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 249.0 |
| 13 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 249.0 | 0.0 | 0.0 |
| 14 | 0.0 | 0.0 | 0.0 | 0.0 | 2.0 | 197.0 | 0.0 | 50.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 15 | 0.0 | 0.0 | 2.0 | 0.0 | 142.0 | 0.0 | 55.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 48.0 | 0.0 |
| 16 | 0.0 | 144.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 105.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 17 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 246.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 18 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 246.0 | 0.0 | 0.0 |
| 19 | 0.0 | 0.0 | 0.0 | 0.0 | 18.0 | 213.0 | 0.0 | 18.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 20 | 0.0 | 5.0 | 1.0 | 12.0 | 15.0 | 27.0 | 3.0 | 168.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 4.0 | 4.0 | 10.0 |
| 21 | 5.0 | 3.0 | 2.0 | 5.0 | 27.0 | 1.0 | 2.0 | 6.0 | 4.0 | 1.0 | 3.0 | 0.0 | 146.0 | 6.0 | 25.0 | 13.0 |
| 22 | 25.0 | 5.0 | 146.0 | 6.0 | 4.0 | 4.0 | 3.0 | 0.0 | 6.0 | 6.0 | 19.0 | 1.0 | 5.0 | 4.0 | 9.0 | 6.0 |
| 23 | 6.0 | 2.0 | 26.0 | 6.0 | 1.0 | 7.0 | 4.0 | 7.0 | 26.0 | 16.0 | 118.0 | 17.0 | 3.0 | 1.0 | 5.0 | 4.0 |
| 24 | 2.0 | 3.0 | 3.0 | 28.0 | 4.0 | 9.0 | 1.0 | 12.0 | 14.0 | 20.0 | 10.0 | 109.0 | 6.0 | 5.0 | 6.0 | 17.0 |
| 25 | 15.0 | 2.0 | 2.0 | 7.0 | 25.0 | 5.0 | 0.0 | 7.0 | 7.0 | 5.0 | 6.0 | 2.0 | 122.0 | 7.0 | 30.0 | 7.0 |
| 26 | 8.0 | 18.0 | 12.0 | 131.0 | 6.0 | 3.0 | 0.0 | 10.0 | 1.0 | 4.0 | 3.0 | 30.0 | 5.0 | 7.0 | 2.0 | 9.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -1.915 | -1.091 | -1.559 | -0.645 | 0.294 | -1.298 | -0.645 | -1.915 | -0.773 | -3.831 | -0.103 | -1.915 | 0.34 | -0.919 | 2.191 | -1.559 |
| 02 | 0.74 | -0.919 | -0.43 | -1.559 | -0.645 | -1.091 | -3.831 | -1.915 | 2.272 | -1.298 | -1.091 | -1.298 | -0.532 | -2.471 | -1.091 | -3.831 |
| 03 | -1.091 | 2.126 | -0.254 | 1.229 | -1.915 | -0.773 | -3.831 | -0.773 | -1.915 | -0.176 | -1.915 | -1.559 | -1.915 | -0.919 | -3.831 | -2.471 |
| 04 | -3.831 | -0.773 | -2.471 | -1.559 | -1.091 | 2.088 | -2.471 | 0.466 | -3.831 | -0.532 | -1.915 | -1.559 | -1.091 | 1.091 | -1.298 | -0.532 |
| 05 | -3.831 | -0.919 | -2.471 | -1.559 | -1.559 | 2.371 | -3.831 | 0.143 | -3.831 | -0.919 | -2.471 | -2.471 | -3.831 | 0.612 | -1.559 | -0.645 |
| 06 | -1.559 | -3.831 | -3.831 | -3.831 | 2.553 | -2.471 | -2.471 | -1.091 | -1.298 | -3.831 | -2.471 | -3.831 | 0.542 | -2.471 | -2.471 | -2.471 |
| 07 | -0.532 | -3.831 | 2.668 | -3.831 | -3.831 | -3.831 | -1.915 | -3.831 | -2.471 | -3.831 | -1.915 | -3.831 | -3.831 | -3.831 | -0.919 | -3.831 |
| 08 | -3.831 | -0.43 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | 2.711 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 |
| 09 | -3.831 | -3.831 | -3.831 | -3.831 | 2.744 | -3.831 | -2.471 | -2.471 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 |
| 10 | 2.732 | -3.831 | -1.559 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -2.471 | -3.831 | -3.831 | -3.831 | -2.471 | -3.831 | -3.831 | -3.831 |
| 11 | -3.831 | -3.831 | -3.831 | 2.74 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -1.559 | -3.831 | -3.831 | -3.831 | -3.831 |
| 12 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | 2.752 |
| 13 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | 2.752 | -3.831 | -3.831 |
| 14 | -3.831 | -3.831 | -3.831 | -3.831 | -1.915 | 2.518 | -3.831 | 1.152 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 |
| 15 | -3.831 | -3.831 | -1.915 | -3.831 | 2.191 | -3.831 | 1.247 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -1.915 | -3.831 | 1.112 | -3.831 |
| 16 | -3.831 | 2.205 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | 1.89 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 |
| 17 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -1.559 | -3.831 | 2.74 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 |
| 18 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -1.559 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | 2.74 | -3.831 | -3.831 |
| 19 | -3.831 | -3.831 | -3.831 | -3.831 | 0.143 | 2.596 | -3.831 | 0.143 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 |
| 20 | -3.831 | -1.091 | -2.471 | -0.254 | -0.036 | 0.542 | -1.559 | 2.359 | -3.831 | -3.831 | -3.831 | -3.831 | -3.831 | -1.298 | -1.298 | -0.43 |
| 21 | -1.091 | -1.559 | -1.915 | -1.091 | 0.542 | -2.471 | -1.915 | -0.919 | -1.298 | -2.471 | -1.559 | -3.831 | 2.219 | -0.919 | 0.466 | -0.176 |
| 22 | 0.466 | -1.091 | 2.219 | -0.919 | -1.298 | -1.298 | -1.559 | -3.831 | -0.919 | -0.919 | 0.196 | -2.471 | -1.091 | -1.298 | -0.532 | -0.919 |
| 23 | -0.919 | -1.915 | 0.504 | -0.919 | -2.471 | -0.773 | -1.298 | -0.773 | 0.504 | 0.027 | 2.007 | 0.087 | -1.559 | -2.471 | -1.091 | -1.298 |
| 24 | -1.915 | -1.559 | -1.559 | 0.578 | -1.298 | -0.532 | -2.471 | -0.254 | -0.103 | 0.246 | -0.43 | 1.928 | -0.919 | -1.091 | -0.919 | 0.087 |
| 25 | -0.036 | -1.915 | -1.915 | -0.773 | 0.466 | -1.091 | -3.831 | -0.773 | -0.773 | -1.091 | -0.919 | -1.915 | 2.04 | -0.773 | 0.646 | -0.773 |
| 26 | -0.645 | 0.143 | -0.254 | 2.111 | -0.919 | -1.559 | -3.831 | -0.43 | -2.471 | -1.298 | -1.559 | 0.646 | -1.091 | -0.773 | -1.915 | -0.532 |