Model info
| Transcription factor | ZNF260 (GeneCards) | ||||||||
| Model | ZN260_HUMAN.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 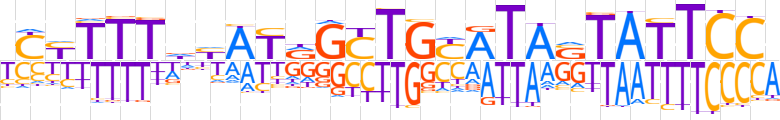 | ||||||||
| LOGO (reverse complement) |  | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 27 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | YCYTTTdYATRGCTGYATASTATTCCA | ||||||||
| Best auROC (human) | 0.937 | ||||||||
| Best auROC (mouse) | |||||||||
| Peak sets in benchmark (human) | 4 | ||||||||
| Peak sets in benchmark (mouse) | |||||||||
| Aligned words | 277 | ||||||||
| TF family | More than 3 adjacent zinc finger factors {2.3.3} | ||||||||
| TF subfamily | ZNF146-like factors {2.3.3.55} | ||||||||
| HGNC | HGNC:13499 | ||||||||
| EntrezGene | GeneID:339324 (SSTAR profile) | ||||||||
| UniProt ID | ZN260_HUMAN | ||||||||
| UniProt AC | Q3ZCT1 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | ZNF260 expression | ||||||||
| ReMap ChIP-seq dataset list | ZNF260 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 2.0 | 3.0 | 0.0 | 0.0 | 15.0 | 48.0 | 1.0 | 3.0 | 0.0 | 7.0 | 0.0 | 1.0 | 7.0 | 161.0 | 5.0 | 23.0 |
| 02 | 1.0 | 1.0 | 2.0 | 20.0 | 14.0 | 73.0 | 1.0 | 131.0 | 0.0 | 3.0 | 2.0 | 1.0 | 3.0 | 11.0 | 2.0 | 11.0 |
| 03 | 1.0 | 2.0 | 0.0 | 15.0 | 8.0 | 6.0 | 3.0 | 71.0 | 0.0 | 0.0 | 0.0 | 7.0 | 2.0 | 4.0 | 4.0 | 153.0 |
| 04 | 1.0 | 3.0 | 0.0 | 7.0 | 1.0 | 1.0 | 0.0 | 10.0 | 0.0 | 0.0 | 2.0 | 5.0 | 2.0 | 7.0 | 7.0 | 230.0 |
| 05 | 1.0 | 1.0 | 0.0 | 2.0 | 1.0 | 3.0 | 1.0 | 6.0 | 1.0 | 1.0 | 1.0 | 6.0 | 1.0 | 3.0 | 12.0 | 236.0 |
| 06 | 1.0 | 2.0 | 1.0 | 0.0 | 2.0 | 0.0 | 3.0 | 3.0 | 3.0 | 2.0 | 0.0 | 9.0 | 89.0 | 19.0 | 39.0 | 103.0 |
| 07 | 7.0 | 33.0 | 1.0 | 54.0 | 5.0 | 5.0 | 8.0 | 5.0 | 7.0 | 8.0 | 2.0 | 26.0 | 10.0 | 31.0 | 5.0 | 69.0 |
| 08 | 24.0 | 2.0 | 1.0 | 2.0 | 58.0 | 8.0 | 4.0 | 7.0 | 15.0 | 0.0 | 0.0 | 1.0 | 139.0 | 4.0 | 2.0 | 9.0 |
| 09 | 2.0 | 64.0 | 1.0 | 169.0 | 0.0 | 3.0 | 0.0 | 11.0 | 0.0 | 0.0 | 0.0 | 7.0 | 0.0 | 7.0 | 1.0 | 11.0 |
| 10 | 0.0 | 0.0 | 1.0 | 1.0 | 35.0 | 6.0 | 16.0 | 17.0 | 0.0 | 1.0 | 0.0 | 1.0 | 17.0 | 2.0 | 148.0 | 31.0 |
| 11 | 0.0 | 0.0 | 52.0 | 0.0 | 5.0 | 0.0 | 3.0 | 1.0 | 14.0 | 1.0 | 143.0 | 7.0 | 2.0 | 2.0 | 45.0 | 1.0 |
| 12 | 0.0 | 4.0 | 0.0 | 17.0 | 0.0 | 2.0 | 0.0 | 1.0 | 0.0 | 185.0 | 2.0 | 56.0 | 1.0 | 4.0 | 0.0 | 4.0 |
| 13 | 0.0 | 0.0 | 1.0 | 0.0 | 2.0 | 2.0 | 0.0 | 191.0 | 0.0 | 1.0 | 0.0 | 1.0 | 1.0 | 3.0 | 0.0 | 74.0 |
| 14 | 0.0 | 2.0 | 1.0 | 0.0 | 2.0 | 1.0 | 0.0 | 3.0 | 0.0 | 0.0 | 1.0 | 0.0 | 2.0 | 18.0 | 243.0 | 3.0 |
| 15 | 4.0 | 0.0 | 0.0 | 0.0 | 1.0 | 13.0 | 0.0 | 7.0 | 22.0 | 169.0 | 1.0 | 53.0 | 2.0 | 2.0 | 0.0 | 2.0 |
| 16 | 14.0 | 0.0 | 14.0 | 1.0 | 162.0 | 3.0 | 19.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 43.0 | 2.0 | 16.0 | 1.0 |
| 17 | 1.0 | 0.0 | 3.0 | 215.0 | 0.0 | 0.0 | 1.0 | 5.0 | 0.0 | 0.0 | 0.0 | 49.0 | 1.0 | 0.0 | 0.0 | 1.0 |
| 18 | 1.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 2.0 | 0.0 | 1.0 | 236.0 | 4.0 | 7.0 | 23.0 |
| 19 | 37.0 | 26.0 | 170.0 | 5.0 | 1.0 | 4.0 | 1.0 | 0.0 | 2.0 | 4.0 | 1.0 | 0.0 | 5.0 | 15.0 | 2.0 | 3.0 |
| 20 | 0.0 | 0.0 | 3.0 | 42.0 | 2.0 | 5.0 | 6.0 | 36.0 | 0.0 | 1.0 | 0.0 | 173.0 | 1.0 | 0.0 | 1.0 | 6.0 |
| 21 | 2.0 | 1.0 | 0.0 | 0.0 | 5.0 | 0.0 | 0.0 | 1.0 | 7.0 | 1.0 | 0.0 | 2.0 | 246.0 | 0.0 | 5.0 | 6.0 |
| 22 | 0.0 | 29.0 | 3.0 | 228.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 1.0 | 0.0 | 4.0 | 0.0 | 3.0 | 0.0 | 6.0 |
| 23 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 32.0 | 0.0 | 1.0 | 0.0 | 2.0 | 0.0 | 1.0 | 2.0 | 237.0 |
| 24 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 | 255.0 | 0.0 | 16.0 |
| 25 | 0.0 | 0.0 | 0.0 | 0.0 | 5.0 | 240.0 | 2.0 | 11.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 13.0 | 1.0 | 4.0 |
| 26 | 0.0 | 3.0 | 0.0 | 2.0 | 222.0 | 6.0 | 7.0 | 18.0 | 0.0 | 2.0 | 0.0 | 1.0 | 8.0 | 0.0 | 7.0 | 0.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -2.013 | -1.659 | -3.914 | -3.914 | -0.137 | 1.011 | -2.567 | -1.659 | -3.914 | -0.873 | -3.914 | -2.567 | -0.873 | 2.216 | -1.191 | 0.283 |
| 02 | -2.567 | -2.567 | -2.013 | 0.145 | -0.204 | 1.427 | -2.567 | 2.01 | -3.914 | -1.659 | -2.013 | -2.567 | -1.659 | -0.439 | -2.013 | -0.439 |
| 03 | -2.567 | -2.013 | -3.914 | -0.137 | -0.746 | -1.019 | -1.659 | 1.4 | -3.914 | -3.914 | -3.914 | -0.873 | -2.013 | -1.398 | -1.398 | 2.165 |
| 04 | -2.567 | -1.659 | -3.914 | -0.873 | -2.567 | -2.567 | -3.914 | -0.531 | -3.914 | -3.914 | -2.013 | -1.191 | -2.013 | -0.873 | -0.873 | 2.572 |
| 05 | -2.567 | -2.567 | -3.914 | -2.013 | -2.567 | -1.659 | -2.567 | -1.019 | -2.567 | -2.567 | -2.567 | -1.019 | -2.567 | -1.659 | -0.354 | 2.597 |
| 06 | -2.567 | -2.013 | -2.567 | -3.914 | -2.013 | -3.914 | -1.659 | -1.659 | -1.659 | -2.013 | -3.914 | -0.632 | 1.625 | 0.095 | 0.805 | 1.77 |
| 07 | -0.873 | 0.639 | -2.567 | 1.127 | -1.191 | -1.191 | -0.746 | -1.191 | -0.873 | -0.746 | -2.013 | 0.404 | -0.531 | 0.577 | -1.191 | 1.371 |
| 08 | 0.325 | -2.013 | -2.567 | -2.013 | 1.199 | -0.746 | -1.398 | -0.873 | -0.137 | -3.914 | -3.914 | -2.567 | 2.069 | -1.398 | -2.013 | -0.632 |
| 09 | -2.013 | 1.296 | -2.567 | 2.264 | -3.914 | -1.659 | -3.914 | -0.439 | -3.914 | -3.914 | -3.914 | -0.873 | -3.914 | -0.873 | -2.567 | -0.439 |
| 10 | -3.914 | -3.914 | -2.567 | -2.567 | 0.697 | -1.019 | -0.074 | -0.014 | -3.914 | -2.567 | -3.914 | -2.567 | -0.014 | -2.013 | 2.132 | 0.577 |
| 11 | -3.914 | -3.914 | 1.09 | -3.914 | -1.191 | -3.914 | -1.659 | -2.567 | -0.204 | -2.567 | 2.097 | -0.873 | -2.013 | -2.013 | 0.946 | -2.567 |
| 12 | -3.914 | -1.398 | -3.914 | -0.014 | -3.914 | -2.013 | -3.914 | -2.567 | -3.914 | 2.354 | -2.013 | 1.164 | -2.567 | -1.398 | -3.914 | -1.398 |
| 13 | -3.914 | -3.914 | -2.567 | -3.914 | -2.013 | -2.013 | -3.914 | 2.386 | -3.914 | -2.567 | -3.914 | -2.567 | -2.567 | -1.659 | -3.914 | 1.441 |
| 14 | -3.914 | -2.013 | -2.567 | -3.914 | -2.013 | -2.567 | -3.914 | -1.659 | -3.914 | -3.914 | -2.567 | -3.914 | -2.013 | 0.042 | 2.627 | -1.659 |
| 15 | -1.398 | -3.914 | -3.914 | -3.914 | -2.567 | -0.276 | -3.914 | -0.873 | 0.239 | 2.264 | -2.567 | 1.109 | -2.013 | -2.013 | -3.914 | -2.013 |
| 16 | -0.204 | -3.914 | -0.204 | -2.567 | 2.222 | -1.659 | 0.095 | -3.914 | -3.914 | -2.567 | -3.914 | -3.914 | 0.901 | -2.013 | -0.074 | -2.567 |
| 17 | -2.567 | -3.914 | -1.659 | 2.504 | -3.914 | -3.914 | -2.567 | -1.191 | -3.914 | -3.914 | -3.914 | 1.031 | -2.567 | -3.914 | -3.914 | -2.567 |
| 18 | -2.567 | -3.914 | -3.914 | -2.567 | -3.914 | -3.914 | -3.914 | -3.914 | -2.567 | -2.013 | -3.914 | -2.567 | 2.597 | -1.398 | -0.873 | 0.283 |
| 19 | 0.752 | 0.404 | 2.27 | -1.191 | -2.567 | -1.398 | -2.567 | -3.914 | -2.013 | -1.398 | -2.567 | -3.914 | -1.191 | -0.137 | -2.013 | -1.659 |
| 20 | -3.914 | -3.914 | -1.659 | 0.878 | -2.013 | -1.191 | -1.019 | 0.725 | -3.914 | -2.567 | -3.914 | 2.287 | -2.567 | -3.914 | -2.567 | -1.019 |
| 21 | -2.013 | -2.567 | -3.914 | -3.914 | -1.191 | -3.914 | -3.914 | -2.567 | -0.873 | -2.567 | -3.914 | -2.013 | 2.639 | -3.914 | -1.191 | -1.019 |
| 22 | -3.914 | 0.511 | -1.659 | 2.563 | -3.914 | -3.914 | -3.914 | -2.013 | -3.914 | -2.567 | -3.914 | -1.398 | -3.914 | -1.659 | -3.914 | -1.019 |
| 23 | -3.914 | -3.914 | -3.914 | -3.914 | -2.567 | -3.914 | -3.914 | 0.609 | -3.914 | -2.567 | -3.914 | -2.013 | -3.914 | -2.567 | -2.013 | 2.602 |
| 24 | -3.914 | -2.567 | -3.914 | -3.914 | -3.914 | -2.567 | -3.914 | -2.567 | -3.914 | -2.567 | -3.914 | -2.567 | -3.914 | 2.675 | -3.914 | -0.074 |
| 25 | -3.914 | -3.914 | -3.914 | -3.914 | -1.191 | 2.614 | -2.013 | -0.439 | -3.914 | -3.914 | -3.914 | -3.914 | -3.914 | -0.276 | -2.567 | -1.398 |
| 26 | -3.914 | -1.659 | -3.914 | -2.013 | 2.536 | -1.019 | -0.873 | 0.042 | -3.914 | -2.013 | -3.914 | -2.567 | -0.746 | -3.914 | -0.873 | -3.914 |