Model info
| Transcription factor | ZNF264 (GeneCards) | ||||||||
| Model | ZN264_HUMAN.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 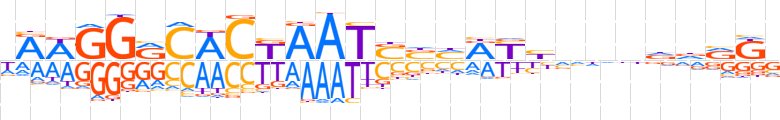 | ||||||||
| LOGO (reverse complement) |  | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 27 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | hRWGGRCWCKAATSbhRYbvdnddRKv | ||||||||
| Best auROC (human) | 0.851 | ||||||||
| Best auROC (mouse) | |||||||||
| Peak sets in benchmark (human) | 2 | ||||||||
| Peak sets in benchmark (mouse) | |||||||||
| Aligned words | 376 | ||||||||
| TF family | More than 3 adjacent zinc finger factors {2.3.3} | ||||||||
| TF subfamily | ZNF460-like factors {2.3.3.54} | ||||||||
| HGNC | HGNC:13057 | ||||||||
| EntrezGene | GeneID:9422 (SSTAR profile) | ||||||||
| UniProt ID | ZN264_HUMAN | ||||||||
| UniProt AC | O43296 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | ZNF264 expression | ||||||||
| ReMap ChIP-seq dataset list | ZNF264 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 86.0 | 9.0 | 11.0 | 4.0 | 29.0 | 9.0 | 1.0 | 5.0 | 20.0 | 3.0 | 9.0 | 6.0 | 158.0 | 5.0 | 16.0 | 4.0 |
| 02 | 212.0 | 5.0 | 17.0 | 59.0 | 16.0 | 3.0 | 2.0 | 5.0 | 30.0 | 4.0 | 1.0 | 2.0 | 10.0 | 3.0 | 6.0 | 0.0 |
| 03 | 28.0 | 5.0 | 231.0 | 4.0 | 6.0 | 0.0 | 7.0 | 2.0 | 5.0 | 1.0 | 18.0 | 2.0 | 2.0 | 1.0 | 62.0 | 1.0 |
| 04 | 5.0 | 1.0 | 34.0 | 1.0 | 1.0 | 1.0 | 4.0 | 1.0 | 5.0 | 1.0 | 298.0 | 14.0 | 0.0 | 0.0 | 9.0 | 0.0 |
| 05 | 3.0 | 3.0 | 3.0 | 2.0 | 1.0 | 2.0 | 0.0 | 0.0 | 101.0 | 29.0 | 209.0 | 6.0 | 3.0 | 0.0 | 13.0 | 0.0 |
| 06 | 36.0 | 69.0 | 2.0 | 1.0 | 3.0 | 28.0 | 0.0 | 3.0 | 5.0 | 217.0 | 1.0 | 2.0 | 1.0 | 5.0 | 1.0 | 1.0 |
| 07 | 12.0 | 0.0 | 2.0 | 31.0 | 267.0 | 3.0 | 19.0 | 30.0 | 2.0 | 0.0 | 2.0 | 0.0 | 4.0 | 2.0 | 0.0 | 1.0 |
| 08 | 0.0 | 263.0 | 16.0 | 6.0 | 0.0 | 5.0 | 0.0 | 0.0 | 0.0 | 21.0 | 2.0 | 0.0 | 0.0 | 50.0 | 12.0 | 0.0 |
| 09 | 0.0 | 0.0 | 0.0 | 0.0 | 13.0 | 21.0 | 58.0 | 247.0 | 4.0 | 3.0 | 11.0 | 12.0 | 0.0 | 3.0 | 1.0 | 2.0 |
| 10 | 11.0 | 0.0 | 6.0 | 0.0 | 18.0 | 4.0 | 3.0 | 2.0 | 68.0 | 1.0 | 0.0 | 1.0 | 229.0 | 6.0 | 17.0 | 9.0 |
| 11 | 316.0 | 4.0 | 3.0 | 3.0 | 9.0 | 0.0 | 0.0 | 2.0 | 18.0 | 4.0 | 0.0 | 4.0 | 10.0 | 1.0 | 0.0 | 1.0 |
| 12 | 2.0 | 40.0 | 5.0 | 306.0 | 0.0 | 0.0 | 1.0 | 8.0 | 0.0 | 0.0 | 0.0 | 3.0 | 2.0 | 2.0 | 2.0 | 4.0 |
| 13 | 2.0 | 1.0 | 1.0 | 0.0 | 3.0 | 19.0 | 5.0 | 15.0 | 0.0 | 5.0 | 3.0 | 0.0 | 6.0 | 220.0 | 60.0 | 35.0 |
| 14 | 0.0 | 6.0 | 1.0 | 4.0 | 22.0 | 180.0 | 5.0 | 38.0 | 6.0 | 5.0 | 57.0 | 1.0 | 6.0 | 28.0 | 7.0 | 9.0 |
| 15 | 8.0 | 12.0 | 3.0 | 11.0 | 15.0 | 152.0 | 3.0 | 49.0 | 52.0 | 9.0 | 4.0 | 5.0 | 2.0 | 33.0 | 6.0 | 11.0 |
| 16 | 62.0 | 5.0 | 9.0 | 1.0 | 163.0 | 16.0 | 9.0 | 18.0 | 4.0 | 3.0 | 4.0 | 5.0 | 45.0 | 4.0 | 19.0 | 8.0 |
| 17 | 5.0 | 22.0 | 23.0 | 224.0 | 3.0 | 11.0 | 5.0 | 9.0 | 6.0 | 9.0 | 3.0 | 23.0 | 6.0 | 9.0 | 8.0 | 9.0 |
| 18 | 4.0 | 7.0 | 3.0 | 6.0 | 15.0 | 9.0 | 0.0 | 27.0 | 5.0 | 19.0 | 10.0 | 5.0 | 7.0 | 111.0 | 19.0 | 128.0 |
| 19 | 7.0 | 4.0 | 16.0 | 4.0 | 99.0 | 19.0 | 20.0 | 8.0 | 4.0 | 6.0 | 19.0 | 3.0 | 16.0 | 87.0 | 34.0 | 29.0 |
| 20 | 10.0 | 11.0 | 20.0 | 85.0 | 81.0 | 15.0 | 10.0 | 10.0 | 27.0 | 11.0 | 37.0 | 14.0 | 12.0 | 10.0 | 18.0 | 4.0 |
| 21 | 22.0 | 15.0 | 10.0 | 83.0 | 11.0 | 13.0 | 7.0 | 16.0 | 33.0 | 10.0 | 21.0 | 21.0 | 6.0 | 43.0 | 47.0 | 17.0 |
| 22 | 9.0 | 9.0 | 20.0 | 34.0 | 38.0 | 9.0 | 27.0 | 7.0 | 44.0 | 6.0 | 25.0 | 10.0 | 17.0 | 5.0 | 103.0 | 12.0 |
| 23 | 30.0 | 4.0 | 49.0 | 25.0 | 11.0 | 5.0 | 5.0 | 8.0 | 121.0 | 8.0 | 28.0 | 18.0 | 8.0 | 21.0 | 17.0 | 17.0 |
| 24 | 35.0 | 8.0 | 121.0 | 6.0 | 28.0 | 4.0 | 2.0 | 4.0 | 10.0 | 9.0 | 72.0 | 8.0 | 17.0 | 25.0 | 22.0 | 4.0 |
| 25 | 6.0 | 8.0 | 52.0 | 24.0 | 4.0 | 4.0 | 36.0 | 2.0 | 22.0 | 10.0 | 176.0 | 9.0 | 0.0 | 3.0 | 15.0 | 4.0 |
| 26 | 12.0 | 3.0 | 14.0 | 3.0 | 6.0 | 12.0 | 3.0 | 4.0 | 36.0 | 60.0 | 166.0 | 17.0 | 2.0 | 3.0 | 31.0 | 3.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 1.289 | -0.932 | -0.739 | -1.695 | 0.21 | -0.932 | -2.855 | -1.489 | -0.156 | -1.955 | -0.932 | -1.318 | 1.895 | -1.489 | -0.375 | -1.695 |
| 02 | 2.188 | -1.489 | -0.315 | 0.914 | -0.375 | -1.955 | -2.307 | -1.489 | 0.243 | -1.695 | -2.855 | -2.307 | -0.831 | -1.955 | -1.318 | -4.163 |
| 03 | 0.175 | -1.489 | 2.274 | -1.695 | -1.318 | -4.163 | -1.173 | -2.307 | -1.489 | -2.855 | -0.259 | -2.307 | -2.307 | -2.855 | 0.963 | -2.855 |
| 04 | -1.489 | -2.855 | 0.367 | -2.855 | -2.855 | -2.855 | -1.695 | -2.855 | -1.489 | -2.855 | 2.528 | -0.505 | -4.163 | -4.163 | -0.932 | -4.163 |
| 05 | -1.955 | -1.955 | -1.955 | -2.307 | -2.855 | -2.307 | -4.163 | -4.163 | 1.449 | 0.21 | 2.174 | -1.318 | -1.955 | -4.163 | -0.577 | -4.163 |
| 06 | 0.424 | 1.069 | -2.307 | -2.855 | -1.955 | 0.175 | -4.163 | -1.955 | -1.489 | 2.212 | -2.855 | -2.307 | -2.855 | -1.489 | -2.855 | -2.855 |
| 07 | -0.655 | -4.163 | -2.307 | 0.276 | 2.419 | -1.955 | -0.206 | 0.243 | -2.307 | -4.163 | -2.307 | -4.163 | -1.695 | -2.307 | -4.163 | -2.855 |
| 08 | -4.163 | 2.404 | -0.375 | -1.318 | -4.163 | -1.489 | -4.163 | -4.163 | -4.163 | -0.108 | -2.307 | -4.163 | -4.163 | 0.749 | -0.655 | -4.163 |
| 09 | -4.163 | -4.163 | -4.163 | -4.163 | -0.577 | -0.108 | 0.897 | 2.341 | -1.695 | -1.955 | -0.739 | -0.655 | -4.163 | -1.955 | -2.855 | -2.307 |
| 10 | -0.739 | -4.163 | -1.318 | -4.163 | -0.259 | -1.695 | -1.955 | -2.307 | 1.055 | -2.855 | -4.163 | -2.855 | 2.265 | -1.318 | -0.315 | -0.932 |
| 11 | 2.587 | -1.695 | -1.955 | -1.955 | -0.932 | -4.163 | -4.163 | -2.307 | -0.259 | -1.695 | -4.163 | -1.695 | -0.831 | -2.855 | -4.163 | -2.855 |
| 12 | -2.307 | 0.528 | -1.489 | 2.555 | -4.163 | -4.163 | -2.855 | -1.045 | -4.163 | -4.163 | -4.163 | -1.955 | -2.307 | -2.307 | -2.307 | -1.695 |
| 13 | -2.307 | -2.855 | -2.855 | -4.163 | -1.955 | -0.206 | -1.489 | -0.438 | -4.163 | -1.489 | -1.955 | -4.163 | -1.318 | 2.225 | 0.93 | 0.396 |
| 14 | -4.163 | -1.318 | -2.855 | -1.695 | -0.062 | 2.025 | -1.489 | 0.477 | -1.318 | -1.489 | 0.88 | -2.855 | -1.318 | 0.175 | -1.173 | -0.932 |
| 15 | -1.045 | -0.655 | -1.955 | -0.739 | -0.438 | 1.856 | -1.955 | 0.729 | 0.788 | -0.932 | -1.695 | -1.489 | -2.307 | 0.338 | -1.318 | -0.739 |
| 16 | 0.963 | -1.489 | -0.932 | -2.855 | 1.926 | -0.375 | -0.932 | -0.259 | -1.695 | -1.955 | -1.695 | -1.489 | 0.645 | -1.695 | -0.206 | -1.045 |
| 17 | -1.489 | -0.062 | -0.019 | 2.243 | -1.955 | -0.739 | -1.489 | -0.932 | -1.318 | -0.932 | -1.955 | -0.019 | -1.318 | -0.932 | -1.045 | -0.932 |
| 18 | -1.695 | -1.173 | -1.955 | -1.318 | -0.438 | -0.932 | -4.163 | 0.139 | -1.489 | -0.206 | -0.831 | -1.489 | -1.173 | 1.543 | -0.206 | 1.685 |
| 19 | -1.173 | -1.695 | -0.375 | -1.695 | 1.429 | -0.206 | -0.156 | -1.045 | -1.695 | -1.318 | -0.206 | -1.955 | -0.375 | 1.3 | 0.367 | 0.21 |
| 20 | -0.831 | -0.739 | -0.156 | 1.277 | 1.229 | -0.438 | -0.831 | -0.831 | 0.139 | -0.739 | 0.451 | -0.505 | -0.655 | -0.831 | -0.259 | -1.695 |
| 21 | -0.062 | -0.438 | -0.831 | 1.253 | -0.739 | -0.577 | -1.173 | -0.375 | 0.338 | -0.831 | -0.108 | -0.108 | -1.318 | 0.6 | 0.688 | -0.315 |
| 22 | -0.932 | -0.932 | -0.156 | 0.367 | 0.477 | -0.932 | 0.139 | -1.173 | 0.623 | -1.318 | 0.064 | -0.831 | -0.315 | -1.489 | 1.468 | -0.655 |
| 23 | 0.243 | -1.695 | 0.729 | 0.064 | -0.739 | -1.489 | -1.489 | -1.045 | 1.629 | -1.045 | 0.175 | -0.259 | -1.045 | -0.108 | -0.315 | -0.315 |
| 24 | 0.396 | -1.045 | 1.629 | -1.318 | 0.175 | -1.695 | -2.307 | -1.695 | -0.831 | -0.932 | 1.112 | -1.045 | -0.315 | 0.064 | -0.062 | -1.695 |
| 25 | -1.318 | -1.045 | 0.788 | 0.023 | -1.695 | -1.695 | 0.424 | -2.307 | -0.062 | -0.831 | 2.003 | -0.932 | -4.163 | -1.955 | -0.438 | -1.695 |
| 26 | -0.655 | -1.955 | -0.505 | -1.955 | -1.318 | -0.655 | -1.955 | -1.695 | 0.424 | 0.93 | 1.944 | -0.315 | -2.307 | -1.955 | 0.276 | -1.955 |