Model info
| Transcription factor | ZNF281 (GeneCards) | ||||||||
| Model | ZN281_HUMAN.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 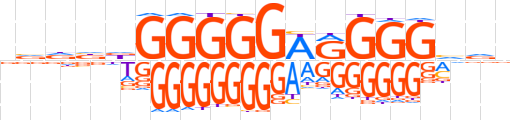 | ||||||||
| LOGO (reverse complement) |  | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 18 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | vvdndGGGGGhRGGGvvd | ||||||||
| Best auROC (human) | 0.986 | ||||||||
| Best auROC (mouse) | 0.978 | ||||||||
| Peak sets in benchmark (human) | 7 | ||||||||
| Peak sets in benchmark (mouse) | 3 | ||||||||
| Aligned words | 561 | ||||||||
| TF family | More than 3 adjacent zinc finger factors {2.3.3} | ||||||||
| TF subfamily | ZNF148-like factors {2.3.3.13} | ||||||||
| HGNC | HGNC:13075 | ||||||||
| EntrezGene | GeneID:23528 (SSTAR profile) | ||||||||
| UniProt ID | ZN281_HUMAN | ||||||||
| UniProt AC | Q9Y2X9 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | ZNF281 expression | ||||||||
| ReMap ChIP-seq dataset list | ZNF281 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 53.0 | 8.0 | 60.0 | 8.0 | 13.0 | 28.0 | 30.0 | 21.0 | 38.0 | 44.0 | 110.0 | 25.0 | 5.0 | 9.0 | 38.0 | 8.0 |
| 02 | 22.0 | 3.0 | 47.0 | 37.0 | 19.0 | 13.0 | 42.0 | 15.0 | 54.0 | 32.0 | 134.0 | 18.0 | 7.0 | 11.0 | 40.0 | 4.0 |
| 03 | 14.0 | 7.0 | 68.0 | 13.0 | 10.0 | 14.0 | 19.0 | 16.0 | 49.0 | 53.0 | 122.0 | 39.0 | 3.0 | 2.0 | 58.0 | 11.0 |
| 04 | 17.0 | 0.0 | 21.0 | 38.0 | 7.0 | 3.0 | 19.0 | 47.0 | 64.0 | 39.0 | 43.0 | 121.0 | 4.0 | 1.0 | 35.0 | 39.0 |
| 05 | 4.0 | 0.0 | 88.0 | 0.0 | 0.0 | 0.0 | 42.0 | 1.0 | 33.0 | 0.0 | 83.0 | 2.0 | 0.0 | 1.0 | 244.0 | 0.0 |
| 06 | 32.0 | 1.0 | 2.0 | 2.0 | 0.0 | 0.0 | 1.0 | 0.0 | 1.0 | 1.0 | 454.0 | 1.0 | 0.0 | 0.0 | 3.0 | 0.0 |
| 07 | 0.0 | 0.0 | 2.0 | 31.0 | 1.0 | 0.0 | 0.0 | 1.0 | 4.0 | 0.0 | 448.0 | 8.0 | 0.0 | 0.0 | 3.0 | 0.0 |
| 08 | 1.0 | 0.0 | 4.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 2.0 | 2.0 | 440.0 | 9.0 | 0.0 | 13.0 | 19.0 | 8.0 |
| 09 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 14.0 | 1.0 | 0.0 | 0.0 | 462.0 | 1.0 | 1.0 | 2.0 | 12.0 | 2.0 |
| 10 | 0.0 | 1.0 | 0.0 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 | 376.0 | 57.0 | 0.0 | 58.0 | 0.0 | 1.0 | 1.0 | 2.0 |
| 11 | 82.0 | 1.0 | 271.0 | 23.0 | 22.0 | 0.0 | 28.0 | 10.0 | 0.0 | 0.0 | 0.0 | 1.0 | 5.0 | 0.0 | 49.0 | 6.0 |
| 12 | 0.0 | 0.0 | 76.0 | 33.0 | 0.0 | 0.0 | 0.0 | 1.0 | 4.0 | 2.0 | 340.0 | 2.0 | 1.0 | 0.0 | 39.0 | 0.0 |
| 13 | 0.0 | 0.0 | 5.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 26.0 | 4.0 | 403.0 | 22.0 | 0.0 | 0.0 | 34.0 | 2.0 |
| 14 | 4.0 | 1.0 | 20.0 | 1.0 | 2.0 | 1.0 | 0.0 | 1.0 | 15.0 | 4.0 | 404.0 | 21.0 | 2.0 | 0.0 | 22.0 | 0.0 |
| 15 | 3.0 | 3.0 | 16.0 | 1.0 | 2.0 | 1.0 | 2.0 | 1.0 | 184.0 | 152.0 | 65.0 | 45.0 | 9.0 | 2.0 | 10.0 | 2.0 |
| 16 | 73.0 | 21.0 | 90.0 | 14.0 | 47.0 | 33.0 | 60.0 | 18.0 | 33.0 | 13.0 | 33.0 | 14.0 | 6.0 | 10.0 | 29.0 | 4.0 |
| 17 | 22.0 | 21.0 | 72.0 | 44.0 | 22.0 | 19.0 | 21.0 | 15.0 | 48.0 | 42.0 | 97.0 | 25.0 | 8.0 | 9.0 | 22.0 | 11.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 0.527 | -1.324 | 0.65 | -1.324 | -0.856 | -0.104 | -0.036 | -0.388 | 0.197 | 0.343 | 1.254 | -0.216 | -1.766 | -1.211 | 0.197 | -1.324 |
| 02 | -0.342 | -2.23 | 0.408 | 0.171 | -0.486 | -0.856 | 0.296 | -0.717 | 0.546 | 0.027 | 1.45 | -0.539 | -1.451 | -1.018 | 0.248 | -1.971 |
| 03 | -0.784 | -1.451 | 0.775 | -0.856 | -1.11 | -0.784 | -0.486 | -0.654 | 0.449 | 0.527 | 1.357 | 0.223 | -2.23 | -2.58 | 0.617 | -1.018 |
| 04 | -0.595 | -4.397 | -0.388 | 0.197 | -1.451 | -2.23 | -0.486 | 0.408 | 0.715 | 0.223 | 0.32 | 1.349 | -1.971 | -3.122 | 0.116 | 0.223 |
| 05 | -1.971 | -4.397 | 1.031 | -4.397 | -4.397 | -4.397 | 0.296 | -3.122 | 0.058 | -4.397 | 0.973 | -2.58 | -4.397 | -3.122 | 2.048 | -4.397 |
| 06 | 0.027 | -3.122 | -2.58 | -2.58 | -4.397 | -4.397 | -3.122 | -4.397 | -3.122 | -3.122 | 2.669 | -3.122 | -4.397 | -4.397 | -2.23 | -4.397 |
| 07 | -4.397 | -4.397 | -2.58 | -0.004 | -3.122 | -4.397 | -4.397 | -3.122 | -1.971 | -4.397 | 2.655 | -1.324 | -4.397 | -4.397 | -2.23 | -4.397 |
| 08 | -3.122 | -4.397 | -1.971 | -4.397 | -4.397 | -4.397 | -4.397 | -4.397 | -2.58 | -2.58 | 2.637 | -1.211 | -4.397 | -0.856 | -0.486 | -1.324 |
| 09 | -4.397 | -4.397 | -2.23 | -4.397 | -4.397 | -4.397 | -0.784 | -3.122 | -4.397 | -4.397 | 2.686 | -3.122 | -3.122 | -2.58 | -0.934 | -2.58 |
| 10 | -4.397 | -3.122 | -4.397 | -4.397 | -3.122 | -3.122 | -4.397 | -4.397 | 2.48 | 0.599 | -4.397 | 0.617 | -4.397 | -3.122 | -3.122 | -2.58 |
| 11 | 0.961 | -3.122 | 2.153 | -0.298 | -0.342 | -4.397 | -0.104 | -1.11 | -4.397 | -4.397 | -4.397 | -3.122 | -1.766 | -4.397 | 0.449 | -1.596 |
| 12 | -4.397 | -4.397 | 0.885 | 0.058 | -4.397 | -4.397 | -4.397 | -3.122 | -1.971 | -2.58 | 2.38 | -2.58 | -3.122 | -4.397 | 0.223 | -4.397 |
| 13 | -4.397 | -4.397 | -1.766 | -4.397 | -4.397 | -4.397 | -2.58 | -4.397 | -0.177 | -1.971 | 2.549 | -0.342 | -4.397 | -4.397 | 0.087 | -2.58 |
| 14 | -1.971 | -3.122 | -0.435 | -3.122 | -2.58 | -3.122 | -4.397 | -3.122 | -0.717 | -1.971 | 2.552 | -0.388 | -2.58 | -4.397 | -0.342 | -4.397 |
| 15 | -2.23 | -2.23 | -0.654 | -3.122 | -2.58 | -3.122 | -2.58 | -3.122 | 1.767 | 1.576 | 0.73 | 0.365 | -1.211 | -2.58 | -1.11 | -2.58 |
| 16 | 0.845 | -0.388 | 1.054 | -0.784 | 0.408 | 0.058 | 0.65 | -0.539 | 0.058 | -0.856 | 0.058 | -0.784 | -1.596 | -1.11 | -0.07 | -1.971 |
| 17 | -0.342 | -0.388 | 0.832 | 0.343 | -0.342 | -0.486 | -0.388 | -0.717 | 0.429 | 0.296 | 1.128 | -0.216 | -1.324 | -1.211 | -0.342 | -1.018 |