Model info
| Transcription factor | ZNF329 (GeneCards) | ||||||||
| Model | ZN329_HUMAN.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 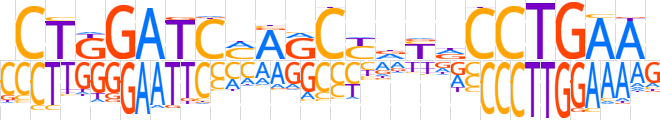 | ||||||||
| LOGO (reverse complement) | 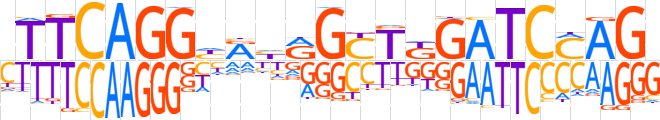 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 23 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | SCTKGATCMARCYdYdCCTGAAR | ||||||||
| Best auROC (human) | 0.857 | ||||||||
| Best auROC (mouse) | |||||||||
| Peak sets in benchmark (human) | 4 | ||||||||
| Peak sets in benchmark (mouse) | |||||||||
| Aligned words | 305 | ||||||||
| TF family | More than 3 adjacent zinc finger factors {2.3.3} | ||||||||
| TF subfamily | unclassified {2.3.3.0} | ||||||||
| HGNC | HGNC:14209 | ||||||||
| EntrezGene | GeneID:79673 (SSTAR profile) | ||||||||
| UniProt ID | ZN329_HUMAN | ||||||||
| UniProt AC | Q86UD4 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | ZNF329 expression | ||||||||
| ReMap ChIP-seq dataset list | ZNF329 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 2.0 | 9.0 | 1.0 | 0.0 | 1.0 | 234.0 | 0.0 | 1.0 | 1.0 | 37.0 | 0.0 | 1.0 | 0.0 | 15.0 | 0.0 | 0.0 |
| 02 | 0.0 | 1.0 | 0.0 | 3.0 | 2.0 | 30.0 | 0.0 | 263.0 | 0.0 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 | 0.0 | 1.0 |
| 03 | 0.0 | 0.0 | 2.0 | 1.0 | 6.0 | 2.0 | 6.0 | 17.0 | 0.0 | 0.0 | 1.0 | 0.0 | 19.0 | 1.0 | 220.0 | 27.0 |
| 04 | 0.0 | 0.0 | 25.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 4.0 | 1.0 | 223.0 | 1.0 | 0.0 | 0.0 | 45.0 | 0.0 |
| 05 | 4.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 287.0 | 2.0 | 4.0 | 3.0 | 1.0 | 0.0 | 0.0 | 0.0 |
| 06 | 6.0 | 3.0 | 16.0 | 267.0 | 0.0 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 | 2.0 | 2.0 | 0.0 | 0.0 | 2.0 | 2.0 |
| 07 | 0.0 | 4.0 | 0.0 | 2.0 | 0.0 | 3.0 | 0.0 | 0.0 | 1.0 | 19.0 | 1.0 | 0.0 | 7.0 | 250.0 | 7.0 | 8.0 |
| 08 | 0.0 | 7.0 | 0.0 | 1.0 | 95.0 | 157.0 | 9.0 | 15.0 | 0.0 | 7.0 | 0.0 | 1.0 | 0.0 | 9.0 | 1.0 | 0.0 |
| 09 | 68.0 | 11.0 | 10.0 | 6.0 | 169.0 | 5.0 | 1.0 | 5.0 | 6.0 | 1.0 | 3.0 | 0.0 | 11.0 | 0.0 | 5.0 | 1.0 |
| 10 | 36.0 | 6.0 | 207.0 | 5.0 | 10.0 | 2.0 | 5.0 | 0.0 | 3.0 | 5.0 | 11.0 | 0.0 | 3.0 | 2.0 | 6.0 | 1.0 |
| 11 | 1.0 | 50.0 | 0.0 | 1.0 | 0.0 | 14.0 | 0.0 | 1.0 | 3.0 | 222.0 | 0.0 | 4.0 | 0.0 | 6.0 | 0.0 | 0.0 |
| 12 | 2.0 | 1.0 | 1.0 | 0.0 | 12.0 | 173.0 | 2.0 | 105.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 5.0 |
| 13 | 3.0 | 2.0 | 7.0 | 3.0 | 116.0 | 16.0 | 18.0 | 24.0 | 1.0 | 0.0 | 2.0 | 0.0 | 32.0 | 4.0 | 67.0 | 7.0 |
| 14 | 4.0 | 20.0 | 16.0 | 112.0 | 3.0 | 2.0 | 1.0 | 16.0 | 11.0 | 12.0 | 8.0 | 63.0 | 1.0 | 9.0 | 2.0 | 22.0 |
| 15 | 9.0 | 1.0 | 7.0 | 2.0 | 26.0 | 0.0 | 7.0 | 10.0 | 15.0 | 2.0 | 8.0 | 2.0 | 68.0 | 7.0 | 108.0 | 30.0 |
| 16 | 1.0 | 113.0 | 1.0 | 3.0 | 0.0 | 9.0 | 0.0 | 1.0 | 2.0 | 127.0 | 0.0 | 1.0 | 0.0 | 43.0 | 0.0 | 1.0 |
| 17 | 1.0 | 2.0 | 0.0 | 0.0 | 1.0 | 289.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 | 1.0 | 1.0 | 4.0 | 1.0 | 0.0 |
| 18 | 0.0 | 0.0 | 1.0 | 2.0 | 1.0 | 0.0 | 1.0 | 293.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 3.0 |
| 19 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 2.0 | 0.0 | 2.0 | 0.0 | 296.0 | 0.0 |
| 20 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 271.0 | 24.0 | 2.0 | 2.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 21 | 249.0 | 1.0 | 8.0 | 16.0 | 19.0 | 1.0 | 2.0 | 2.0 | 2.0 | 0.0 | 0.0 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 |
| 22 | 42.0 | 20.0 | 194.0 | 15.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 1.0 | 8.0 | 1.0 | 1.0 | 3.0 | 13.0 | 1.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -2.099 | -0.72 | -2.651 | -3.987 | -2.651 | 2.5 | -3.987 | -2.651 | -2.651 | 0.664 | -3.987 | -2.651 | -3.987 | -0.225 | -3.987 | -3.987 |
| 02 | -3.987 | -2.651 | -3.987 | -1.746 | -2.099 | 0.456 | -3.987 | 2.617 | -3.987 | -3.987 | -2.651 | -3.987 | -2.651 | -3.987 | -3.987 | -2.651 |
| 03 | -3.987 | -3.987 | -2.099 | -2.651 | -1.107 | -2.099 | -1.107 | -0.103 | -3.987 | -3.987 | -2.651 | -3.987 | 0.006 | -2.651 | 2.439 | 0.352 |
| 04 | -3.987 | -3.987 | 0.276 | -3.987 | -3.987 | -3.987 | -1.746 | -3.987 | -1.485 | -2.651 | 2.452 | -2.651 | -3.987 | -3.987 | 0.858 | -3.987 |
| 05 | -1.485 | -3.987 | -3.987 | -3.987 | -3.987 | -3.987 | -3.987 | -2.651 | 2.704 | -2.099 | -1.485 | -1.746 | -2.651 | -3.987 | -3.987 | -3.987 |
| 06 | -1.107 | -1.746 | -0.162 | 2.632 | -3.987 | -3.987 | -2.651 | -2.651 | -3.987 | -3.987 | -2.099 | -2.099 | -3.987 | -3.987 | -2.099 | -2.099 |
| 07 | -3.987 | -1.485 | -3.987 | -2.099 | -3.987 | -1.746 | -3.987 | -3.987 | -2.651 | 0.006 | -2.651 | -3.987 | -0.961 | 2.566 | -0.961 | -0.833 |
| 08 | -3.987 | -0.961 | -3.987 | -2.651 | 1.601 | 2.102 | -0.72 | -0.225 | -3.987 | -0.961 | -3.987 | -2.651 | -3.987 | -0.72 | -2.651 | -3.987 |
| 09 | 1.268 | -0.527 | -0.619 | -1.107 | 2.175 | -1.278 | -2.651 | -1.278 | -1.107 | -2.651 | -1.746 | -3.987 | -0.527 | -3.987 | -1.278 | -2.651 |
| 10 | 0.637 | -1.107 | 2.378 | -1.278 | -0.619 | -2.099 | -1.278 | -3.987 | -1.746 | -1.278 | -0.527 | -3.987 | -1.746 | -2.099 | -1.107 | -2.651 |
| 11 | -2.651 | 0.963 | -3.987 | -2.651 | -3.987 | -0.292 | -3.987 | -2.651 | -1.746 | 2.448 | -3.987 | -1.485 | -3.987 | -1.107 | -3.987 | -3.987 |
| 12 | -2.099 | -2.651 | -2.651 | -3.987 | -0.442 | 2.199 | -2.099 | 1.701 | -3.987 | -3.987 | -3.987 | -3.987 | -2.651 | -3.987 | -3.987 | -1.278 |
| 13 | -1.746 | -2.099 | -0.961 | -1.746 | 1.8 | -0.162 | -0.047 | 0.236 | -2.651 | -3.987 | -2.099 | -3.987 | 0.52 | -1.485 | 1.253 | -0.961 |
| 14 | -1.485 | 0.057 | -0.162 | 1.765 | -1.746 | -2.099 | -2.651 | -0.162 | -0.527 | -0.442 | -0.833 | 1.192 | -2.651 | -0.72 | -2.099 | 0.151 |
| 15 | -0.72 | -2.651 | -0.961 | -2.099 | 0.315 | -3.987 | -0.961 | -0.619 | -0.225 | -2.099 | -0.833 | -2.099 | 1.268 | -0.961 | 1.729 | 0.456 |
| 16 | -2.651 | 1.774 | -2.651 | -1.746 | -3.987 | -0.72 | -3.987 | -2.651 | -2.099 | 1.89 | -3.987 | -2.651 | -3.987 | 0.813 | -3.987 | -2.651 |
| 17 | -2.651 | -2.099 | -3.987 | -3.987 | -2.651 | 2.711 | -3.987 | -2.099 | -3.987 | -3.987 | -3.987 | -2.651 | -2.651 | -1.485 | -2.651 | -3.987 |
| 18 | -3.987 | -3.987 | -2.651 | -2.099 | -2.651 | -3.987 | -2.651 | 2.725 | -3.987 | -3.987 | -2.651 | -3.987 | -3.987 | -3.987 | -3.987 | -1.746 |
| 19 | -3.987 | -3.987 | -2.651 | -3.987 | -3.987 | -3.987 | -3.987 | -3.987 | -2.651 | -3.987 | -2.099 | -3.987 | -2.099 | -3.987 | 2.735 | -3.987 |
| 20 | -1.746 | -3.987 | -3.987 | -3.987 | -3.987 | -3.987 | -3.987 | -3.987 | 2.647 | 0.236 | -2.099 | -2.099 | -3.987 | -3.987 | -3.987 | -3.987 |
| 21 | 2.562 | -2.651 | -0.833 | -0.162 | 0.006 | -2.651 | -2.099 | -2.099 | -2.099 | -3.987 | -3.987 | -3.987 | -2.651 | -2.651 | -3.987 | -3.987 |
| 22 | 0.79 | 0.057 | 2.313 | -0.225 | -3.987 | -3.987 | -3.987 | -1.746 | -3.987 | -2.651 | -0.833 | -2.651 | -2.651 | -1.746 | -0.365 | -2.651 |