Model info
| Transcription factor | ZNF382 (GeneCards) | ||||||||
| Model | ZN382_HUMAN.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 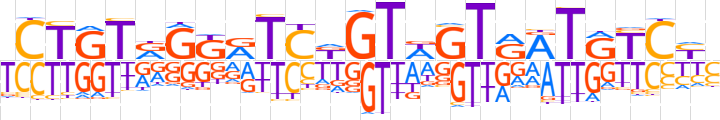 | ||||||||
| LOGO (reverse complement) | 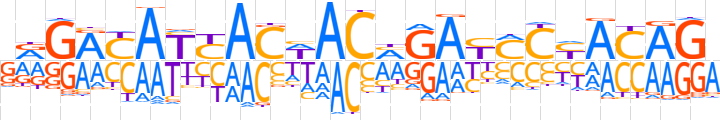 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 25 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | TCTGTRGKRTCKGTWGTRATRTCYC | ||||||||
| Best auROC (human) | 0.922 | ||||||||
| Best auROC (mouse) | |||||||||
| Peak sets in benchmark (human) | 2 | ||||||||
| Peak sets in benchmark (mouse) | |||||||||
| Aligned words | 302 | ||||||||
| TF family | Factors with multiple dispersed zinc fingers {2.3.4} | ||||||||
| TF subfamily | ZNF37A-like factors {2.3.4.25} | ||||||||
| HGNC | HGNC:17409 | ||||||||
| EntrezGene | GeneID:84911 (SSTAR profile) | ||||||||
| UniProt ID | ZN382_HUMAN | ||||||||
| UniProt AC | Q96SR6 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | ZNF382 expression | ||||||||
| ReMap ChIP-seq dataset list | ZNF382 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 3.0 | 9.0 | 1.0 | 1.0 | 0.0 | 17.0 | 1.0 | 0.0 | 0.0 | 16.0 | 0.0 | 3.0 | 6.0 | 233.0 | 4.0 | 8.0 |
| 02 | 0.0 | 2.0 | 0.0 | 7.0 | 4.0 | 31.0 | 1.0 | 239.0 | 0.0 | 3.0 | 1.0 | 2.0 | 1.0 | 1.0 | 1.0 | 9.0 |
| 03 | 2.0 | 1.0 | 2.0 | 0.0 | 24.0 | 1.0 | 9.0 | 3.0 | 0.0 | 0.0 | 3.0 | 0.0 | 20.0 | 1.0 | 231.0 | 5.0 |
| 04 | 2.0 | 2.0 | 3.0 | 39.0 | 1.0 | 0.0 | 0.0 | 2.0 | 2.0 | 15.0 | 1.0 | 227.0 | 2.0 | 3.0 | 0.0 | 3.0 |
| 05 | 3.0 | 0.0 | 2.0 | 2.0 | 14.0 | 0.0 | 2.0 | 4.0 | 1.0 | 0.0 | 0.0 | 3.0 | 118.0 | 1.0 | 127.0 | 25.0 |
| 06 | 18.0 | 0.0 | 112.0 | 6.0 | 1.0 | 0.0 | 0.0 | 0.0 | 10.0 | 0.0 | 115.0 | 6.0 | 3.0 | 0.0 | 27.0 | 4.0 |
| 07 | 2.0 | 2.0 | 22.0 | 6.0 | 0.0 | 0.0 | 0.0 | 0.0 | 9.0 | 6.0 | 187.0 | 52.0 | 2.0 | 1.0 | 11.0 | 2.0 |
| 08 | 7.0 | 2.0 | 4.0 | 0.0 | 7.0 | 0.0 | 2.0 | 0.0 | 115.0 | 7.0 | 95.0 | 3.0 | 33.0 | 1.0 | 25.0 | 1.0 |
| 09 | 0.0 | 11.0 | 0.0 | 151.0 | 1.0 | 1.0 | 0.0 | 8.0 | 4.0 | 6.0 | 1.0 | 115.0 | 0.0 | 0.0 | 0.0 | 4.0 |
| 10 | 0.0 | 5.0 | 0.0 | 0.0 | 0.0 | 15.0 | 1.0 | 2.0 | 0.0 | 1.0 | 0.0 | 0.0 | 3.0 | 226.0 | 4.0 | 45.0 |
| 11 | 0.0 | 1.0 | 0.0 | 2.0 | 54.0 | 7.0 | 15.0 | 171.0 | 1.0 | 0.0 | 2.0 | 2.0 | 0.0 | 1.0 | 40.0 | 6.0 |
| 12 | 0.0 | 0.0 | 54.0 | 1.0 | 2.0 | 0.0 | 6.0 | 1.0 | 0.0 | 0.0 | 57.0 | 0.0 | 10.0 | 2.0 | 169.0 | 0.0 |
| 13 | 0.0 | 0.0 | 0.0 | 12.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 2.0 | 1.0 | 283.0 | 0.0 | 0.0 | 0.0 | 2.0 |
| 14 | 0.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 168.0 | 0.0 | 54.0 | 77.0 |
| 15 | 6.0 | 17.0 | 146.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 3.0 | 3.0 | 47.0 | 1.0 | 7.0 | 0.0 | 67.0 | 4.0 |
| 16 | 0.0 | 1.0 | 0.0 | 15.0 | 0.0 | 0.0 | 0.0 | 20.0 | 1.0 | 2.0 | 0.0 | 257.0 | 0.0 | 0.0 | 0.0 | 6.0 |
| 17 | 0.0 | 1.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 118.0 | 2.0 | 174.0 | 4.0 |
| 18 | 92.0 | 8.0 | 16.0 | 5.0 | 1.0 | 0.0 | 1.0 | 1.0 | 147.0 | 6.0 | 12.0 | 9.0 | 3.0 | 0.0 | 0.0 | 1.0 |
| 19 | 0.0 | 3.0 | 0.0 | 240.0 | 0.0 | 1.0 | 0.0 | 13.0 | 0.0 | 1.0 | 1.0 | 27.0 | 1.0 | 3.0 | 0.0 | 12.0 |
| 20 | 0.0 | 0.0 | 0.0 | 1.0 | 4.0 | 2.0 | 0.0 | 2.0 | 0.0 | 0.0 | 1.0 | 0.0 | 60.0 | 1.0 | 221.0 | 10.0 |
| 21 | 4.0 | 3.0 | 10.0 | 47.0 | 1.0 | 1.0 | 0.0 | 1.0 | 4.0 | 19.0 | 8.0 | 191.0 | 1.0 | 1.0 | 3.0 | 8.0 |
| 22 | 0.0 | 10.0 | 0.0 | 0.0 | 0.0 | 18.0 | 0.0 | 6.0 | 0.0 | 21.0 | 0.0 | 0.0 | 8.0 | 222.0 | 5.0 | 12.0 |
| 23 | 0.0 | 5.0 | 0.0 | 3.0 | 37.0 | 92.0 | 0.0 | 142.0 | 1.0 | 2.0 | 0.0 | 2.0 | 2.0 | 8.0 | 1.0 | 7.0 |
| 24 | 1.0 | 28.0 | 3.0 | 8.0 | 4.0 | 90.0 | 0.0 | 13.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 142.0 | 2.0 | 10.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -1.746 | -0.72 | -2.651 | -2.651 | -3.987 | -0.103 | -2.651 | -3.987 | -3.987 | -0.162 | -3.987 | -1.746 | -1.107 | 2.496 | -1.485 | -0.833 |
| 02 | -3.987 | -2.099 | -3.987 | -0.961 | -1.485 | 0.489 | -2.651 | 2.521 | -3.987 | -1.746 | -2.651 | -2.099 | -2.651 | -2.651 | -2.651 | -0.72 |
| 03 | -2.099 | -2.651 | -2.099 | -3.987 | 0.236 | -2.651 | -0.72 | -1.746 | -3.987 | -3.987 | -1.746 | -3.987 | 0.057 | -2.651 | 2.487 | -1.278 |
| 04 | -2.099 | -2.099 | -1.746 | 0.716 | -2.651 | -3.987 | -3.987 | -2.099 | -2.099 | -0.225 | -2.651 | 2.47 | -2.099 | -1.746 | -3.987 | -1.746 |
| 05 | -1.746 | -3.987 | -2.099 | -2.099 | -0.292 | -3.987 | -2.099 | -1.485 | -2.651 | -3.987 | -3.987 | -1.746 | 1.817 | -2.651 | 1.89 | 0.276 |
| 06 | -0.047 | -3.987 | 1.765 | -1.107 | -2.651 | -3.987 | -3.987 | -3.987 | -0.619 | -3.987 | 1.791 | -1.107 | -1.746 | -3.987 | 0.352 | -1.485 |
| 07 | -2.099 | -2.099 | 0.151 | -1.107 | -3.987 | -3.987 | -3.987 | -3.987 | -0.72 | -1.107 | 2.276 | 1.002 | -2.099 | -2.651 | -0.527 | -2.099 |
| 08 | -0.961 | -2.099 | -1.485 | -3.987 | -0.961 | -3.987 | -2.099 | -3.987 | 1.791 | -0.961 | 1.601 | -1.746 | 0.551 | -2.651 | 0.276 | -2.651 |
| 09 | -3.987 | -0.527 | -3.987 | 2.063 | -2.651 | -2.651 | -3.987 | -0.833 | -1.485 | -1.107 | -2.651 | 1.791 | -3.987 | -3.987 | -3.987 | -1.485 |
| 10 | -3.987 | -1.278 | -3.987 | -3.987 | -3.987 | -0.225 | -2.651 | -2.099 | -3.987 | -2.651 | -3.987 | -3.987 | -1.746 | 2.466 | -1.485 | 0.858 |
| 11 | -3.987 | -2.651 | -3.987 | -2.099 | 1.039 | -0.961 | -0.225 | 2.187 | -2.651 | -3.987 | -2.099 | -2.099 | -3.987 | -2.651 | 0.741 | -1.107 |
| 12 | -3.987 | -3.987 | 1.039 | -2.651 | -2.099 | -3.987 | -1.107 | -2.651 | -3.987 | -3.987 | 1.093 | -3.987 | -0.619 | -2.099 | 2.175 | -3.987 |
| 13 | -3.987 | -3.987 | -3.987 | -0.442 | -3.987 | -3.987 | -3.987 | -2.099 | -3.987 | -2.099 | -2.651 | 2.69 | -3.987 | -3.987 | -3.987 | -2.099 |
| 14 | -3.987 | -3.987 | -3.987 | -3.987 | -2.099 | -3.987 | -3.987 | -3.987 | -3.987 | -3.987 | -3.987 | -2.651 | 2.17 | -3.987 | 1.039 | 1.392 |
| 15 | -1.107 | -0.103 | 2.029 | -2.651 | -3.987 | -3.987 | -3.987 | -3.987 | -1.746 | -1.746 | 0.901 | -2.651 | -0.961 | -3.987 | 1.253 | -1.485 |
| 16 | -3.987 | -2.651 | -3.987 | -0.225 | -3.987 | -3.987 | -3.987 | 0.057 | -2.651 | -2.099 | -3.987 | 2.594 | -3.987 | -3.987 | -3.987 | -1.107 |
| 17 | -3.987 | -2.651 | -3.987 | -3.987 | -1.746 | -3.987 | -3.987 | -3.987 | -3.987 | -3.987 | -3.987 | -3.987 | 1.817 | -2.099 | 2.205 | -1.485 |
| 18 | 1.569 | -0.833 | -0.162 | -1.278 | -2.651 | -3.987 | -2.651 | -2.651 | 2.036 | -1.107 | -0.442 | -0.72 | -1.746 | -3.987 | -3.987 | -2.651 |
| 19 | -3.987 | -1.746 | -3.987 | 2.526 | -3.987 | -2.651 | -3.987 | -0.365 | -3.987 | -2.651 | -2.651 | 0.352 | -2.651 | -1.746 | -3.987 | -0.442 |
| 20 | -3.987 | -3.987 | -3.987 | -2.651 | -1.485 | -2.099 | -3.987 | -2.099 | -3.987 | -3.987 | -2.651 | -3.987 | 1.144 | -2.651 | 2.443 | -0.619 |
| 21 | -1.485 | -1.746 | -0.619 | 0.901 | -2.651 | -2.651 | -3.987 | -2.651 | -1.485 | 0.006 | -0.833 | 2.298 | -2.651 | -2.651 | -1.746 | -0.833 |
| 22 | -3.987 | -0.619 | -3.987 | -3.987 | -3.987 | -0.047 | -3.987 | -1.107 | -3.987 | 0.105 | -3.987 | -3.987 | -0.833 | 2.448 | -1.278 | -0.442 |
| 23 | -3.987 | -1.278 | -3.987 | -1.746 | 0.664 | 1.569 | -3.987 | 2.002 | -2.651 | -2.099 | -3.987 | -2.099 | -2.099 | -0.833 | -2.651 | -0.961 |
| 24 | -2.651 | 0.388 | -1.746 | -0.833 | -1.485 | 1.547 | -3.987 | -0.365 | -3.987 | -2.651 | -3.987 | -3.987 | -3.987 | 2.002 | -2.099 | -0.619 |