Model info
| Transcription factor | ZNF436 (GeneCards) | ||||||||
| Model | ZN436_HUMAN.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 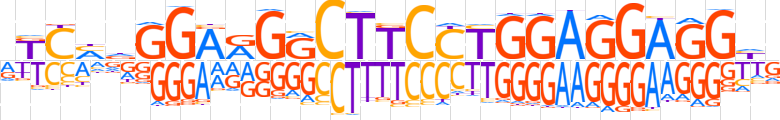 | ||||||||
| LOGO (reverse complement) | 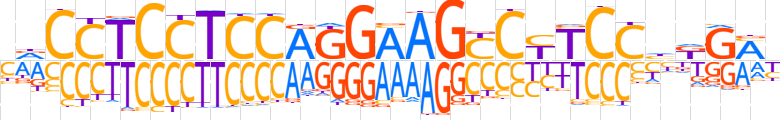 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 27 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | RKCMdGGARGRCTTCCTGGAGGAGGhG | ||||||||
| Best auROC (human) | 0.984 | ||||||||
| Best auROC (mouse) | |||||||||
| Peak sets in benchmark (human) | 4 | ||||||||
| Peak sets in benchmark (mouse) | |||||||||
| Aligned words | 485 | ||||||||
| TF family | More than 3 adjacent zinc finger factors {2.3.3} | ||||||||
| TF subfamily | ZNF180-like factors {2.3.3.58} | ||||||||
| HGNC | HGNC:20814 | ||||||||
| EntrezGene | GeneID:80818 (SSTAR profile) | ||||||||
| UniProt ID | ZN436_HUMAN | ||||||||
| UniProt AC | Q9C0F3 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | ZNF436 expression | ||||||||
| ReMap ChIP-seq dataset list | ZNF436 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 16.0 | 25.0 | 35.0 | 215.0 | 2.0 | 3.0 | 0.0 | 7.0 | 7.0 | 6.0 | 20.0 | 120.0 | 2.0 | 4.0 | 2.0 | 21.0 |
| 02 | 2.0 | 9.0 | 1.0 | 15.0 | 0.0 | 33.0 | 0.0 | 5.0 | 5.0 | 33.0 | 5.0 | 14.0 | 3.0 | 339.0 | 12.0 | 9.0 |
| 03 | 3.0 | 4.0 | 1.0 | 2.0 | 242.0 | 113.0 | 3.0 | 56.0 | 9.0 | 8.0 | 1.0 | 0.0 | 4.0 | 30.0 | 8.0 | 1.0 |
| 04 | 57.0 | 14.0 | 169.0 | 18.0 | 75.0 | 28.0 | 11.0 | 41.0 | 3.0 | 1.0 | 5.0 | 4.0 | 11.0 | 4.0 | 40.0 | 4.0 |
| 05 | 7.0 | 1.0 | 138.0 | 0.0 | 13.0 | 5.0 | 20.0 | 9.0 | 20.0 | 1.0 | 202.0 | 2.0 | 2.0 | 2.0 | 62.0 | 1.0 |
| 06 | 2.0 | 0.0 | 40.0 | 0.0 | 1.0 | 1.0 | 7.0 | 0.0 | 9.0 | 0.0 | 411.0 | 2.0 | 0.0 | 0.0 | 12.0 | 0.0 |
| 07 | 10.0 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 | 0.0 | 1.0 | 400.0 | 24.0 | 31.0 | 15.0 | 2.0 | 0.0 | 0.0 | 0.0 |
| 08 | 206.0 | 1.0 | 202.0 | 3.0 | 16.0 | 4.0 | 3.0 | 1.0 | 24.0 | 1.0 | 4.0 | 3.0 | 3.0 | 0.0 | 14.0 | 0.0 |
| 09 | 6.0 | 2.0 | 239.0 | 2.0 | 2.0 | 0.0 | 4.0 | 0.0 | 11.0 | 1.0 | 211.0 | 0.0 | 0.0 | 0.0 | 7.0 | 0.0 |
| 10 | 1.0 | 2.0 | 16.0 | 0.0 | 1.0 | 0.0 | 2.0 | 0.0 | 79.0 | 26.0 | 348.0 | 8.0 | 0.0 | 1.0 | 1.0 | 0.0 |
| 11 | 0.0 | 81.0 | 0.0 | 0.0 | 0.0 | 29.0 | 0.0 | 0.0 | 0.0 | 367.0 | 0.0 | 0.0 | 0.0 | 8.0 | 0.0 | 0.0 |
| 12 | 0.0 | 0.0 | 0.0 | 0.0 | 10.0 | 12.0 | 5.0 | 458.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 13 | 1.0 | 0.0 | 7.0 | 2.0 | 1.0 | 0.0 | 0.0 | 11.0 | 0.0 | 1.0 | 3.0 | 1.0 | 3.0 | 26.0 | 32.0 | 397.0 |
| 14 | 0.0 | 5.0 | 0.0 | 0.0 | 0.0 | 27.0 | 0.0 | 0.0 | 0.0 | 40.0 | 1.0 | 1.0 | 0.0 | 403.0 | 1.0 | 7.0 |
| 15 | 0.0 | 0.0 | 0.0 | 0.0 | 34.0 | 401.0 | 5.0 | 35.0 | 1.0 | 1.0 | 0.0 | 0.0 | 1.0 | 6.0 | 0.0 | 1.0 |
| 16 | 0.0 | 6.0 | 4.0 | 26.0 | 14.0 | 33.0 | 2.0 | 359.0 | 0.0 | 2.0 | 0.0 | 3.0 | 2.0 | 9.0 | 3.0 | 22.0 |
| 17 | 1.0 | 0.0 | 15.0 | 0.0 | 18.0 | 0.0 | 21.0 | 11.0 | 1.0 | 0.0 | 7.0 | 1.0 | 5.0 | 0.0 | 404.0 | 1.0 |
| 18 | 1.0 | 0.0 | 24.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 15.0 | 7.0 | 414.0 | 11.0 | 0.0 | 0.0 | 12.0 | 1.0 |
| 19 | 13.0 | 0.0 | 1.0 | 2.0 | 7.0 | 0.0 | 0.0 | 0.0 | 430.0 | 2.0 | 7.0 | 11.0 | 12.0 | 0.0 | 0.0 | 0.0 |
| 20 | 54.0 | 0.0 | 408.0 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 | 2.0 | 0.0 | 6.0 | 0.0 | 0.0 | 0.0 | 13.0 | 0.0 |
| 21 | 1.0 | 0.0 | 55.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 4.0 | 1.0 | 423.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 22 | 5.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 435.0 | 17.0 | 6.0 | 20.0 | 1.0 | 0.0 | 0.0 | 0.0 |
| 23 | 69.0 | 0.0 | 372.0 | 0.0 | 6.0 | 0.0 | 11.0 | 0.0 | 1.0 | 0.0 | 5.0 | 0.0 | 0.0 | 0.0 | 21.0 | 0.0 |
| 24 | 2.0 | 0.0 | 74.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 11.0 | 1.0 | 391.0 | 6.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 25 | 0.0 | 1.0 | 7.0 | 5.0 | 0.0 | 0.0 | 0.0 | 1.0 | 78.0 | 104.0 | 22.0 | 261.0 | 2.0 | 1.0 | 1.0 | 2.0 |
| 26 | 11.0 | 4.0 | 65.0 | 0.0 | 61.0 | 8.0 | 35.0 | 2.0 | 6.0 | 2.0 | 22.0 | 0.0 | 11.0 | 4.0 | 251.0 | 3.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -0.628 | -0.19 | 0.142 | 1.948 | -2.554 | -2.204 | -4.375 | -1.425 | -1.425 | -1.57 | -0.409 | 1.366 | -2.554 | -1.946 | -2.554 | -0.361 |
| 02 | -2.554 | -1.185 | -3.097 | -0.691 | -4.375 | 0.084 | -4.375 | -1.74 | -1.74 | 0.084 | -1.74 | -0.758 | -2.204 | 2.403 | -0.908 | -1.185 |
| 03 | -2.204 | -1.946 | -3.097 | -2.554 | 2.066 | 1.307 | -2.204 | 0.608 | -1.185 | -1.298 | -3.097 | -4.375 | -1.946 | -0.01 | -1.298 | -3.097 |
| 04 | 0.626 | -0.758 | 1.708 | -0.513 | 0.898 | -0.078 | -0.992 | 0.299 | -2.204 | -3.097 | -1.74 | -1.946 | -0.992 | -1.946 | 0.274 | -1.946 |
| 05 | -1.425 | -3.097 | 1.506 | -4.375 | -0.83 | -1.74 | -0.409 | -1.185 | -0.409 | -3.097 | 1.886 | -2.554 | -2.554 | -2.554 | 0.709 | -3.097 |
| 06 | -2.554 | -4.375 | 0.274 | -4.375 | -3.097 | -3.097 | -1.425 | -4.375 | -1.185 | -4.375 | 2.595 | -2.554 | -4.375 | -4.375 | -0.908 | -4.375 |
| 07 | -1.084 | -4.375 | -3.097 | -3.097 | -4.375 | -4.375 | -4.375 | -3.097 | 2.568 | -0.23 | 0.022 | -0.691 | -2.554 | -4.375 | -4.375 | -4.375 |
| 08 | 1.906 | -3.097 | 1.886 | -2.204 | -0.628 | -1.946 | -2.204 | -3.097 | -0.23 | -3.097 | -1.946 | -2.204 | -2.204 | -4.375 | -0.758 | -4.375 |
| 09 | -1.57 | -2.554 | 2.054 | -2.554 | -2.554 | -4.375 | -1.946 | -4.375 | -0.992 | -3.097 | 1.929 | -4.375 | -4.375 | -4.375 | -1.425 | -4.375 |
| 10 | -3.097 | -2.554 | -0.628 | -4.375 | -3.097 | -4.375 | -2.554 | -4.375 | 0.95 | -0.151 | 2.429 | -1.298 | -4.375 | -3.097 | -3.097 | -4.375 |
| 11 | -4.375 | 0.975 | -4.375 | -4.375 | -4.375 | -0.044 | -4.375 | -4.375 | -4.375 | 2.482 | -4.375 | -4.375 | -4.375 | -1.298 | -4.375 | -4.375 |
| 12 | -4.375 | -4.375 | -4.375 | -4.375 | -1.084 | -0.908 | -1.74 | 2.703 | -4.375 | -4.375 | -4.375 | -4.375 | -4.375 | -4.375 | -4.375 | -4.375 |
| 13 | -3.097 | -4.375 | -1.425 | -2.554 | -3.097 | -4.375 | -4.375 | -0.992 | -4.375 | -3.097 | -2.204 | -3.097 | -2.204 | -0.151 | 0.054 | 2.561 |
| 14 | -4.375 | -1.74 | -4.375 | -4.375 | -4.375 | -0.114 | -4.375 | -4.375 | -4.375 | 0.274 | -3.097 | -3.097 | -4.375 | 2.576 | -3.097 | -1.425 |
| 15 | -4.375 | -4.375 | -4.375 | -4.375 | 0.113 | 2.571 | -1.74 | 0.142 | -3.097 | -3.097 | -4.375 | -4.375 | -3.097 | -1.57 | -4.375 | -3.097 |
| 16 | -4.375 | -1.57 | -1.946 | -0.151 | -0.758 | 0.084 | -2.554 | 2.46 | -4.375 | -2.554 | -4.375 | -2.204 | -2.554 | -1.185 | -2.204 | -0.316 |
| 17 | -3.097 | -4.375 | -0.691 | -4.375 | -0.513 | -4.375 | -0.361 | -0.992 | -3.097 | -4.375 | -1.425 | -3.097 | -1.74 | -4.375 | 2.578 | -3.097 |
| 18 | -3.097 | -4.375 | -0.23 | -4.375 | -4.375 | -4.375 | -4.375 | -4.375 | -0.691 | -1.425 | 2.603 | -0.992 | -4.375 | -4.375 | -0.908 | -3.097 |
| 19 | -0.83 | -4.375 | -3.097 | -2.554 | -1.425 | -4.375 | -4.375 | -4.375 | 2.64 | -2.554 | -1.425 | -0.992 | -0.908 | -4.375 | -4.375 | -4.375 |
| 20 | 0.572 | -4.375 | 2.588 | -4.375 | -3.097 | -4.375 | -3.097 | -4.375 | -2.554 | -4.375 | -1.57 | -4.375 | -4.375 | -4.375 | -0.83 | -4.375 |
| 21 | -3.097 | -4.375 | 0.59 | -3.097 | -4.375 | -4.375 | -4.375 | -4.375 | -1.946 | -3.097 | 2.624 | -4.375 | -4.375 | -4.375 | -4.375 | -4.375 |
| 22 | -1.74 | -4.375 | -4.375 | -4.375 | -4.375 | -4.375 | -4.375 | -3.097 | 2.652 | -0.569 | -1.57 | -0.409 | -3.097 | -4.375 | -4.375 | -4.375 |
| 23 | 0.815 | -4.375 | 2.496 | -4.375 | -1.57 | -4.375 | -0.992 | -4.375 | -3.097 | -4.375 | -1.74 | -4.375 | -4.375 | -4.375 | -0.361 | -4.375 |
| 24 | -2.554 | -4.375 | 0.885 | -4.375 | -4.375 | -4.375 | -4.375 | -4.375 | -0.992 | -3.097 | 2.545 | -1.57 | -4.375 | -4.375 | -4.375 | -4.375 |
| 25 | -4.375 | -3.097 | -1.425 | -1.74 | -4.375 | -4.375 | -4.375 | -3.097 | 0.937 | 1.224 | -0.316 | 2.142 | -2.554 | -3.097 | -3.097 | -2.554 |
| 26 | -0.992 | -1.946 | 0.756 | -4.375 | 0.693 | -1.298 | 0.142 | -2.554 | -1.57 | -2.554 | -0.316 | -4.375 | -0.992 | -1.946 | 2.103 | -2.204 |