Model info
| Transcription factor | ZNF502 (GeneCards) | ||||||||
| Model | ZN502_HUMAN.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 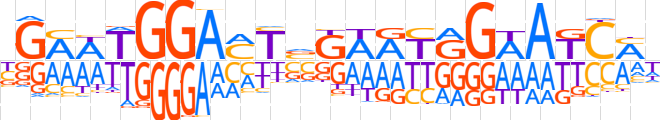 | ||||||||
| LOGO (reverse complement) |  | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 23 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | bGMWTGGAMTvGAATGGWATCMW | ||||||||
| Best auROC (human) | 0.967 | ||||||||
| Best auROC (mouse) | |||||||||
| Peak sets in benchmark (human) | 3 | ||||||||
| Peak sets in benchmark (mouse) | |||||||||
| Aligned words | 189 | ||||||||
| TF family | More than 3 adjacent zinc finger factors {2.3.3} | ||||||||
| TF subfamily | unclassified {2.3.3.0} | ||||||||
| HGNC | HGNC:23718 | ||||||||
| EntrezGene | GeneID:91392 (SSTAR profile) | ||||||||
| UniProt ID | ZN502_HUMAN | ||||||||
| UniProt AC | Q8TBZ5 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | ZNF502 expression | ||||||||
| ReMap ChIP-seq dataset list | ZNF502 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 1.0 | 0.0 | 5.0 | 0.0 | 14.0 | 1.0 | 57.0 | 0.0 | 0.0 | 0.0 | 34.0 | 0.0 | 6.0 | 0.0 | 58.0 | 0.0 |
| 02 | 11.0 | 8.0 | 0.0 | 2.0 | 0.0 | 1.0 | 0.0 | 0.0 | 109.0 | 43.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 03 | 116.0 | 0.0 | 0.0 | 4.0 | 6.0 | 13.0 | 1.0 | 32.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 1.0 |
| 04 | 3.0 | 1.0 | 0.0 | 118.0 | 5.0 | 0.0 | 0.0 | 11.0 | 0.0 | 0.0 | 0.0 | 1.0 | 14.0 | 2.0 | 6.0 | 15.0 |
| 05 | 1.0 | 0.0 | 21.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 6.0 | 0.0 | 0.0 | 0.0 | 145.0 | 0.0 |
| 06 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 175.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 07 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 168.0 | 5.0 | 2.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 08 | 70.0 | 95.0 | 1.0 | 2.0 | 0.0 | 4.0 | 0.0 | 1.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 |
| 09 | 0.0 | 0.0 | 0.0 | 70.0 | 6.0 | 20.0 | 3.0 | 72.0 | 0.0 | 1.0 | 0.0 | 0.0 | 1.0 | 1.0 | 0.0 | 2.0 |
| 10 | 0.0 | 3.0 | 4.0 | 0.0 | 6.0 | 8.0 | 2.0 | 6.0 | 2.0 | 0.0 | 1.0 | 0.0 | 19.0 | 73.0 | 49.0 | 3.0 |
| 11 | 0.0 | 5.0 | 5.0 | 17.0 | 0.0 | 0.0 | 78.0 | 6.0 | 1.0 | 1.0 | 48.0 | 6.0 | 1.0 | 2.0 | 6.0 | 0.0 |
| 12 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 8.0 | 112.0 | 0.0 | 0.0 | 25.0 | 4.0 | 0.0 | 0.0 | 25.0 |
| 13 | 111.0 | 0.0 | 5.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 2.0 | 0.0 | 56.0 | 0.0 |
| 14 | 1.0 | 1.0 | 0.0 | 111.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 60.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 15 | 0.0 | 0.0 | 1.0 | 0.0 | 61.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 5.0 | 0.0 | 109.0 | 0.0 |
| 16 | 2.0 | 0.0 | 64.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 110.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 17 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 | 0.0 | 111.0 | 1.0 | 1.0 | 61.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 18 | 110.0 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 61.0 | 0.0 | 2.0 | 0.0 |
| 19 | 0.0 | 2.0 | 56.0 | 115.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 20 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 0.0 | 5.0 | 44.0 | 3.0 | 7.0 | 0.0 | 112.0 | 3.0 | 0.0 |
| 21 | 0.0 | 4.0 | 0.0 | 1.0 | 116.0 | 28.0 | 4.0 | 10.0 | 1.0 | 5.0 | 0.0 | 0.0 | 1.0 | 6.0 | 0.0 | 0.0 |
| 22 | 30.0 | 8.0 | 4.0 | 76.0 | 14.0 | 1.0 | 0.0 | 28.0 | 3.0 | 0.0 | 0.0 | 1.0 | 5.0 | 3.0 | 0.0 | 3.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -2.147 | -3.556 | -0.755 | -3.556 | 0.235 | -2.147 | 1.622 | -3.556 | -3.556 | -3.556 | 1.109 | -3.556 | -0.583 | -3.556 | 1.639 | -3.556 |
| 02 | 0.0 | -0.308 | -3.556 | -1.584 | -3.556 | -2.147 | -3.556 | -3.556 | 2.267 | 1.342 | -3.556 | -1.584 | -3.556 | -3.556 | -3.556 | -3.556 |
| 03 | 2.33 | -3.556 | -3.556 | -0.963 | -0.583 | 0.163 | -2.147 | 1.049 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | -1.226 | -3.556 | -2.147 |
| 04 | -1.226 | -2.147 | -3.556 | 2.347 | -0.755 | -3.556 | -3.556 | 0.0 | -3.556 | -3.556 | -3.556 | -2.147 | 0.235 | -1.584 | -0.583 | 0.303 |
| 05 | -2.147 | -3.556 | 0.633 | -3.556 | -3.556 | -3.556 | -1.226 | -3.556 | -3.556 | -3.556 | -0.583 | -3.556 | -3.556 | -3.556 | 2.552 | -3.556 |
| 06 | -3.556 | -3.556 | -2.147 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | 2.74 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 |
| 07 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | 2.699 | -0.755 | -1.584 | -2.147 | -3.556 | -3.556 | -3.556 | -3.556 |
| 08 | 1.826 | 2.13 | -2.147 | -1.584 | -3.556 | -0.963 | -3.556 | -2.147 | -3.556 | -1.584 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | -2.147 |
| 09 | -3.556 | -3.556 | -3.556 | 1.826 | -0.583 | 0.585 | -1.226 | 1.854 | -3.556 | -2.147 | -3.556 | -3.556 | -2.147 | -2.147 | -3.556 | -1.584 |
| 10 | -3.556 | -1.226 | -0.963 | -3.556 | -0.583 | -0.308 | -1.584 | -0.583 | -1.584 | -3.556 | -2.147 | -3.556 | 0.534 | 1.868 | 1.472 | -1.226 |
| 11 | -3.556 | -0.755 | -0.755 | 0.425 | -3.556 | -3.556 | 1.934 | -0.583 | -2.147 | -2.147 | 1.451 | -0.583 | -2.147 | -1.584 | -0.583 | -3.556 |
| 12 | -3.556 | -2.147 | -2.147 | -3.556 | -3.556 | -3.556 | -3.556 | -0.308 | 2.295 | -3.556 | -3.556 | 0.805 | -0.963 | -3.556 | -3.556 | 0.805 |
| 13 | 2.286 | -3.556 | -0.755 | -3.556 | -3.556 | -3.556 | -2.147 | -3.556 | -3.556 | -3.556 | -2.147 | -3.556 | -1.584 | -3.556 | 1.604 | -3.556 |
| 14 | -2.147 | -2.147 | -3.556 | 2.286 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | 1.673 | -3.556 | -1.226 | -3.556 | -3.556 | -3.556 | -3.556 |
| 15 | -3.556 | -3.556 | -2.147 | -3.556 | 1.689 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | -0.755 | -3.556 | 2.267 | -3.556 |
| 16 | -1.584 | -3.556 | 1.737 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | 2.277 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 |
| 17 | -3.556 | -3.556 | -3.556 | -1.584 | -3.556 | -3.556 | -3.556 | -3.556 | 2.286 | -2.147 | -2.147 | 1.689 | -3.556 | -3.556 | -3.556 | -3.556 |
| 18 | 2.277 | -3.556 | -2.147 | -3.556 | -2.147 | -3.556 | -3.556 | -3.556 | -2.147 | -3.556 | -3.556 | -3.556 | 1.689 | -3.556 | -1.584 | -3.556 |
| 19 | -3.556 | -1.584 | 1.604 | 2.321 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | -1.226 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 |
| 20 | -3.556 | -3.556 | -3.556 | -3.556 | -3.556 | -1.584 | -3.556 | -3.556 | -0.755 | 1.365 | -1.226 | -0.436 | -3.556 | 2.295 | -1.226 | -3.556 |
| 21 | -3.556 | -0.963 | -3.556 | -2.147 | 2.33 | 0.917 | -0.963 | -0.092 | -2.147 | -0.755 | -3.556 | -3.556 | -2.147 | -0.583 | -3.556 | -3.556 |
| 22 | 0.985 | -0.308 | -0.963 | 1.908 | 0.235 | -2.147 | -3.556 | 0.917 | -1.226 | -3.556 | -3.556 | -2.147 | -0.755 | -1.226 | -3.556 | -1.226 |