Model info
| Transcription factor | ZNF586 (GeneCards) | ||||||||
| Model | ZN586_HUMAN.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 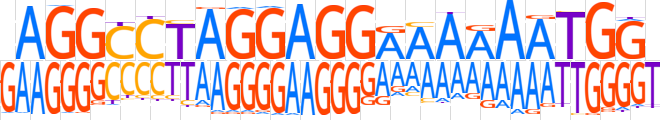 | ||||||||
| LOGO (reverse complement) | 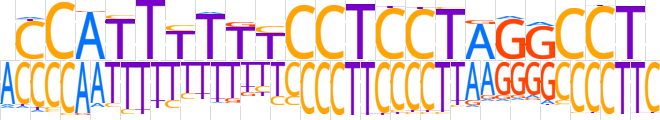 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 23 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | GAGGCCTAGGAGGAAAAAATGGT | ||||||||
| Best auROC (human) | 0.999 | ||||||||
| Best auROC (mouse) | |||||||||
| Peak sets in benchmark (human) | 4 | ||||||||
| Peak sets in benchmark (mouse) | |||||||||
| Aligned words | 481 | ||||||||
| TF family | More than 3 adjacent zinc finger factors {2.3.3} | ||||||||
| TF subfamily | unclassified {2.3.3.0} | ||||||||
| HGNC | HGNC:25949 | ||||||||
| EntrezGene | GeneID:54807 (SSTAR profile) | ||||||||
| UniProt ID | ZN586_HUMAN | ||||||||
| UniProt AC | Q9NXT0 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | ZNF586 expression | ||||||||
| ReMap ChIP-seq dataset list | ZNF586 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 12.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 | 452.0 | 1.0 | 6.0 | 4.0 | 4.0 | 0.0 | 0.0 | 0.0 |
| 02 | 8.0 | 0.0 | 460.0 | 2.0 | 1.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 4.0 | 0.0 | 0.0 | 0.0 | 4.0 | 0.0 |
| 03 | 0.0 | 0.0 | 11.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 7.0 | 5.0 | 452.0 | 4.0 | 0.0 | 0.0 | 2.0 | 0.0 |
| 04 | 0.0 | 5.0 | 1.0 | 1.0 | 0.0 | 2.0 | 0.0 | 3.0 | 11.0 | 403.0 | 9.0 | 42.0 | 0.0 | 4.0 | 0.0 | 0.0 |
| 05 | 0.0 | 10.0 | 0.0 | 1.0 | 7.0 | 385.0 | 3.0 | 19.0 | 0.0 | 9.0 | 0.0 | 1.0 | 2.0 | 32.0 | 1.0 | 11.0 |
| 06 | 0.0 | 1.0 | 0.0 | 8.0 | 5.0 | 50.0 | 2.0 | 379.0 | 0.0 | 0.0 | 0.0 | 4.0 | 2.0 | 1.0 | 3.0 | 26.0 |
| 07 | 6.0 | 0.0 | 0.0 | 1.0 | 51.0 | 0.0 | 1.0 | 0.0 | 5.0 | 0.0 | 0.0 | 0.0 | 400.0 | 1.0 | 16.0 | 0.0 |
| 08 | 18.0 | 0.0 | 443.0 | 1.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 17.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 |
| 09 | 0.0 | 0.0 | 19.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 15.0 | 1.0 | 443.0 | 2.0 | 0.0 | 0.0 | 1.0 | 0.0 |
| 10 | 14.0 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 455.0 | 0.0 | 7.0 | 1.0 | 2.0 | 0.0 | 0.0 | 0.0 |
| 11 | 6.0 | 0.0 | 463.0 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 6.0 | 1.0 | 0.0 | 0.0 | 1.0 | 0.0 |
| 12 | 0.0 | 0.0 | 7.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 7.0 | 0.0 | 459.0 | 4.0 | 0.0 | 0.0 | 4.0 | 0.0 |
| 13 | 5.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 365.0 | 9.0 | 95.0 | 1.0 | 3.0 | 1.0 | 0.0 | 0.0 |
| 14 | 318.0 | 48.0 | 0.0 | 7.0 | 9.0 | 1.0 | 0.0 | 0.0 | 80.0 | 10.0 | 5.0 | 2.0 | 1.0 | 0.0 | 0.0 | 0.0 |
| 15 | 395.0 | 6.0 | 2.0 | 5.0 | 42.0 | 0.0 | 1.0 | 16.0 | 5.0 | 0.0 | 0.0 | 0.0 | 8.0 | 0.0 | 1.0 | 0.0 |
| 16 | 366.0 | 5.0 | 72.0 | 7.0 | 6.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 1.0 | 0.0 | 11.0 | 0.0 | 9.0 | 1.0 |
| 17 | 386.0 | 0.0 | 0.0 | 0.0 | 5.0 | 0.0 | 0.0 | 0.0 | 82.0 | 0.0 | 0.0 | 0.0 | 8.0 | 0.0 | 0.0 | 0.0 |
| 18 | 432.0 | 2.0 | 44.0 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 19 | 6.0 | 10.0 | 2.0 | 414.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 4.0 | 0.0 | 40.0 | 0.0 | 0.0 | 3.0 | 0.0 |
| 20 | 0.0 | 0.0 | 4.0 | 2.0 | 0.0 | 0.0 | 14.0 | 0.0 | 1.0 | 0.0 | 4.0 | 0.0 | 2.0 | 0.0 | 454.0 | 0.0 |
| 21 | 1.0 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 25.0 | 3.0 | 430.0 | 18.0 | 0.0 | 0.0 | 2.0 | 0.0 |
| 22 | 0.0 | 3.0 | 0.0 | 23.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 6.0 | 6.0 | 421.0 | 0.0 | 0.0 | 0.0 | 19.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -0.899 | -4.368 | -4.368 | -4.368 | -2.546 | -4.368 | -4.368 | -4.368 | 2.698 | -3.09 | -1.562 | -1.938 | -1.938 | -4.368 | -4.368 | -4.368 |
| 02 | -1.289 | -4.368 | 2.716 | -2.546 | -3.09 | -4.368 | -4.368 | -4.368 | -2.546 | -4.368 | -1.938 | -4.368 | -4.368 | -4.368 | -1.938 | -4.368 |
| 03 | -4.368 | -4.368 | -0.984 | -4.368 | -4.368 | -4.368 | -4.368 | -4.368 | -1.416 | -1.732 | 2.698 | -1.938 | -4.368 | -4.368 | -2.546 | -4.368 |
| 04 | -4.368 | -1.732 | -3.09 | -3.09 | -4.368 | -2.546 | -4.368 | -2.196 | -0.984 | 2.584 | -1.177 | 0.331 | -4.368 | -1.938 | -4.368 | -4.368 |
| 05 | -4.368 | -1.076 | -4.368 | -3.09 | -1.416 | 2.538 | -2.196 | -0.451 | -4.368 | -1.177 | -4.368 | -3.09 | -2.546 | 0.062 | -3.09 | -0.984 |
| 06 | -4.368 | -3.09 | -4.368 | -1.289 | -1.732 | 0.504 | -2.546 | 2.523 | -4.368 | -4.368 | -4.368 | -1.938 | -2.546 | -3.09 | -2.196 | -0.143 |
| 07 | -1.562 | -4.368 | -4.368 | -3.09 | 0.523 | -4.368 | -3.09 | -4.368 | -1.732 | -4.368 | -4.368 | -4.368 | 2.576 | -3.09 | -0.62 | -4.368 |
| 08 | -0.504 | -4.368 | 2.678 | -3.09 | -3.09 | -4.368 | -4.368 | -4.368 | -4.368 | -4.368 | -0.56 | -4.368 | -4.368 | -4.368 | -3.09 | -4.368 |
| 09 | -4.368 | -4.368 | -0.451 | -4.368 | -4.368 | -4.368 | -4.368 | -4.368 | -0.683 | -3.09 | 2.678 | -2.546 | -4.368 | -4.368 | -3.09 | -4.368 |
| 10 | -0.75 | -4.368 | -3.09 | -4.368 | -3.09 | -4.368 | -4.368 | -4.368 | 2.705 | -4.368 | -1.416 | -3.09 | -2.546 | -4.368 | -4.368 | -4.368 |
| 11 | -1.562 | -4.368 | 2.723 | -2.196 | -4.368 | -4.368 | -4.368 | -4.368 | -3.09 | -4.368 | -1.562 | -3.09 | -4.368 | -4.368 | -3.09 | -4.368 |
| 12 | -4.368 | -4.368 | -1.416 | -4.368 | -4.368 | -4.368 | -4.368 | -4.368 | -1.416 | -4.368 | 2.714 | -1.938 | -4.368 | -4.368 | -1.938 | -4.368 |
| 13 | -1.732 | -4.368 | -2.546 | -4.368 | -4.368 | -4.368 | -4.368 | -4.368 | 2.485 | -1.177 | 1.142 | -3.09 | -2.196 | -3.09 | -4.368 | -4.368 |
| 14 | 2.347 | 0.463 | -4.368 | -1.416 | -1.177 | -3.09 | -4.368 | -4.368 | 0.971 | -1.076 | -1.732 | -2.546 | -3.09 | -4.368 | -4.368 | -4.368 |
| 15 | 2.564 | -1.562 | -2.546 | -1.732 | 0.331 | -4.368 | -3.09 | -0.62 | -1.732 | -4.368 | -4.368 | -4.368 | -1.289 | -4.368 | -3.09 | -4.368 |
| 16 | 2.488 | -1.732 | 0.866 | -1.416 | -1.562 | -4.368 | -4.368 | -4.368 | -2.196 | -4.368 | -3.09 | -4.368 | -0.984 | -4.368 | -1.177 | -3.09 |
| 17 | 2.541 | -4.368 | -4.368 | -4.368 | -1.732 | -4.368 | -4.368 | -4.368 | 0.995 | -4.368 | -4.368 | -4.368 | -1.289 | -4.368 | -4.368 | -4.368 |
| 18 | 2.653 | -2.546 | 0.377 | -2.196 | -4.368 | -4.368 | -4.368 | -4.368 | -4.368 | -4.368 | -4.368 | -4.368 | -4.368 | -4.368 | -4.368 | -4.368 |
| 19 | -1.562 | -1.076 | -2.546 | 2.611 | -4.368 | -4.368 | -4.368 | -2.546 | -4.368 | -1.938 | -4.368 | 0.282 | -4.368 | -4.368 | -2.196 | -4.368 |
| 20 | -4.368 | -4.368 | -1.938 | -2.546 | -4.368 | -4.368 | -0.75 | -4.368 | -3.09 | -4.368 | -1.938 | -4.368 | -2.546 | -4.368 | 2.703 | -4.368 |
| 21 | -3.09 | -4.368 | -3.09 | -3.09 | -4.368 | -4.368 | -4.368 | -4.368 | -0.182 | -2.196 | 2.649 | -0.504 | -4.368 | -4.368 | -2.546 | -4.368 |
| 22 | -4.368 | -2.196 | -4.368 | -0.264 | -4.368 | -4.368 | -4.368 | -2.196 | -4.368 | -1.562 | -1.562 | 2.628 | -4.368 | -4.368 | -4.368 | -0.451 |