Model info
| Transcription factor | ZNF680 (GeneCards) | ||||||||
| Model | ZN680_HUMAN.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 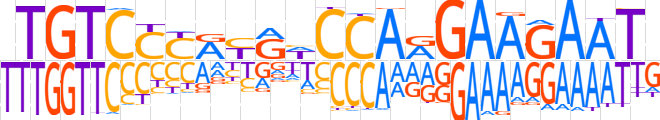 | ||||||||
| LOGO (reverse complement) | 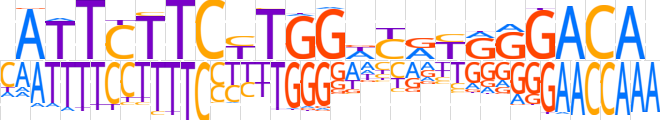 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 23 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | TTGTCCYRYGWCCARGAAGAATR | ||||||||
| Best auROC (human) | 0.989 | ||||||||
| Best auROC (mouse) | |||||||||
| Peak sets in benchmark (human) | 3 | ||||||||
| Peak sets in benchmark (mouse) | |||||||||
| Aligned words | 422 | ||||||||
| TF family | More than 3 adjacent zinc finger factors {2.3.3} | ||||||||
| TF subfamily | unclassified {2.3.3.0} | ||||||||
| HGNC | HGNC:26897 | ||||||||
| EntrezGene | GeneID:340252 (SSTAR profile) | ||||||||
| UniProt ID | ZN680_HUMAN | ||||||||
| UniProt AC | Q8NEM1 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | ZNF680 expression | ||||||||
| ReMap ChIP-seq dataset list | ZNF680 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 1.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 7.0 | 2.0 | 1.0 | 1.0 | 7.0 | 1.0 | 4.0 | 0.0 | 397.0 |
| 02 | 1.0 | 0.0 | 4.0 | 0.0 | 1.0 | 1.0 | 2.0 | 1.0 | 1.0 | 0.0 | 0.0 | 0.0 | 2.0 | 1.0 | 407.0 | 1.0 |
| 03 | 1.0 | 0.0 | 1.0 | 3.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 7.0 | 1.0 | 405.0 | 0.0 | 0.0 | 1.0 | 1.0 |
| 04 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 5.0 | 0.0 | 2.0 | 1.0 | 2.0 | 0.0 | 0.0 | 12.0 | 396.0 | 1.0 | 2.0 |
| 05 | 0.0 | 13.0 | 0.0 | 0.0 | 0.0 | 309.0 | 1.0 | 94.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 3.0 | 1.0 | 0.0 |
| 06 | 0.0 | 0.0 | 0.0 | 0.0 | 16.0 | 226.0 | 1.0 | 82.0 | 0.0 | 2.0 | 1.0 | 0.0 | 0.0 | 85.0 | 7.0 | 2.0 |
| 07 | 0.0 | 1.0 | 14.0 | 1.0 | 266.0 | 5.0 | 34.0 | 8.0 | 0.0 | 0.0 | 9.0 | 0.0 | 9.0 | 1.0 | 74.0 | 0.0 |
| 08 | 5.0 | 124.0 | 7.0 | 139.0 | 0.0 | 4.0 | 0.0 | 3.0 | 3.0 | 54.0 | 2.0 | 72.0 | 1.0 | 5.0 | 1.0 | 2.0 |
| 09 | 0.0 | 0.0 | 9.0 | 0.0 | 132.0 | 1.0 | 53.0 | 1.0 | 3.0 | 0.0 | 7.0 | 0.0 | 11.0 | 0.0 | 205.0 | 0.0 |
| 10 | 40.0 | 15.0 | 7.0 | 84.0 | 0.0 | 0.0 | 0.0 | 1.0 | 78.0 | 33.0 | 11.0 | 152.0 | 0.0 | 1.0 | 0.0 | 0.0 |
| 11 | 1.0 | 113.0 | 0.0 | 4.0 | 0.0 | 47.0 | 0.0 | 2.0 | 0.0 | 17.0 | 0.0 | 1.0 | 0.0 | 227.0 | 1.0 | 9.0 |
| 12 | 0.0 | 1.0 | 0.0 | 0.0 | 12.0 | 374.0 | 0.0 | 18.0 | 0.0 | 1.0 | 0.0 | 0.0 | 1.0 | 14.0 | 1.0 | 0.0 |
| 13 | 10.0 | 1.0 | 2.0 | 0.0 | 384.0 | 2.0 | 4.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 11.0 | 0.0 | 6.0 | 1.0 |
| 14 | 221.0 | 3.0 | 171.0 | 11.0 | 3.0 | 0.0 | 0.0 | 0.0 | 5.0 | 0.0 | 6.0 | 1.0 | 0.0 | 0.0 | 0.0 | 1.0 |
| 15 | 0.0 | 0.0 | 229.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 177.0 | 0.0 | 0.0 | 0.0 | 13.0 | 0.0 |
| 16 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 422.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 17 | 387.0 | 0.0 | 34.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 18 | 46.0 | 1.0 | 336.0 | 4.0 | 0.0 | 0.0 | 0.0 | 0.0 | 4.0 | 0.0 | 26.0 | 4.0 | 0.0 | 0.0 | 1.0 | 0.0 |
| 19 | 50.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 362.0 | 1.0 | 0.0 | 0.0 | 8.0 | 0.0 | 0.0 | 0.0 |
| 20 | 376.0 | 8.0 | 3.0 | 34.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 21 | 2.0 | 7.0 | 2.0 | 366.0 | 1.0 | 0.0 | 0.0 | 7.0 | 0.0 | 0.0 | 1.0 | 2.0 | 0.0 | 2.0 | 0.0 | 32.0 |
| 22 | 0.0 | 0.0 | 3.0 | 0.0 | 6.0 | 1.0 | 0.0 | 2.0 | 1.0 | 0.0 | 2.0 | 0.0 | 62.0 | 8.0 | 282.0 | 55.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -2.966 | -4.26 | -4.26 | -4.26 | -2.966 | -4.26 | -4.26 | -1.288 | -2.42 | -2.966 | -2.966 | -1.288 | -2.966 | -1.81 | -4.26 | 2.698 |
| 02 | -2.966 | -4.26 | -1.81 | -4.26 | -2.966 | -2.966 | -2.42 | -2.966 | -2.966 | -4.26 | -4.26 | -4.26 | -2.42 | -2.966 | 2.723 | -2.966 |
| 03 | -2.966 | -4.26 | -2.966 | -2.069 | -4.26 | -4.26 | -4.26 | -2.42 | -4.26 | -1.288 | -2.966 | 2.718 | -4.26 | -4.26 | -2.966 | -2.966 |
| 04 | -4.26 | -2.966 | -4.26 | -4.26 | -4.26 | -1.604 | -4.26 | -2.42 | -2.966 | -2.42 | -4.26 | -4.26 | -0.771 | 2.696 | -2.966 | -2.42 |
| 05 | -4.26 | -0.693 | -4.26 | -4.26 | -4.26 | 2.448 | -2.966 | 1.261 | -4.26 | -4.26 | -2.966 | -4.26 | -4.26 | -2.069 | -2.966 | -4.26 |
| 06 | -4.26 | -4.26 | -4.26 | -4.26 | -0.491 | 2.136 | -2.966 | 1.125 | -4.26 | -2.42 | -2.966 | -4.26 | -4.26 | 1.16 | -1.288 | -2.42 |
| 07 | -4.26 | -2.966 | -0.621 | -2.966 | 2.298 | -1.604 | 0.251 | -1.161 | -4.26 | -4.26 | -1.048 | -4.26 | -1.048 | -2.966 | 1.023 | -4.26 |
| 08 | -1.604 | 1.537 | -1.288 | 1.651 | -4.26 | -1.81 | -4.26 | -2.069 | -2.069 | 0.709 | -2.42 | 0.995 | -2.966 | -1.604 | -2.966 | -2.42 |
| 09 | -4.26 | -4.26 | -1.048 | -4.26 | 1.599 | -2.966 | 0.691 | -2.966 | -2.069 | -4.26 | -1.288 | -4.26 | -0.855 | -4.26 | 2.038 | -4.26 |
| 10 | 0.412 | -0.554 | -1.288 | 1.149 | -4.26 | -4.26 | -4.26 | -2.966 | 1.075 | 0.221 | -0.855 | 1.74 | -4.26 | -2.966 | -4.26 | -4.26 |
| 11 | -2.966 | 1.444 | -4.26 | -1.81 | -4.26 | 0.572 | -4.26 | -2.42 | -4.26 | -0.431 | -4.26 | -2.966 | -4.26 | 2.14 | -2.966 | -1.048 |
| 12 | -4.26 | -2.966 | -4.26 | -4.26 | -0.771 | 2.639 | -4.26 | -0.375 | -4.26 | -2.966 | -4.26 | -4.26 | -2.966 | -0.621 | -2.966 | -4.26 |
| 13 | -0.947 | -2.966 | -2.42 | -4.26 | 2.665 | -2.42 | -1.81 | -4.26 | -2.966 | -4.26 | -4.26 | -4.26 | -0.855 | -4.26 | -1.434 | -2.966 |
| 14 | 2.113 | -2.069 | 1.857 | -0.855 | -2.069 | -4.26 | -4.26 | -4.26 | -1.604 | -4.26 | -1.434 | -2.966 | -4.26 | -4.26 | -4.26 | -2.966 |
| 15 | -4.26 | -4.26 | 2.149 | -4.26 | -4.26 | -4.26 | -2.069 | -4.26 | -4.26 | -4.26 | 1.892 | -4.26 | -4.26 | -4.26 | -0.693 | -4.26 |
| 16 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | 2.759 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 |
| 17 | 2.673 | -4.26 | 0.251 | -2.966 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 |
| 18 | 0.55 | -2.966 | 2.532 | -1.81 | -4.26 | -4.26 | -4.26 | -4.26 | -1.81 | -4.26 | -0.014 | -1.81 | -4.26 | -4.26 | -2.966 | -4.26 |
| 19 | 0.633 | -4.26 | -4.26 | -4.26 | -2.966 | -4.26 | -4.26 | -4.26 | 2.606 | -2.966 | -4.26 | -4.26 | -1.161 | -4.26 | -4.26 | -4.26 |
| 20 | 2.644 | -1.161 | -2.069 | 0.251 | -2.966 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 | -4.26 |
| 21 | -2.42 | -1.288 | -2.42 | 2.617 | -2.966 | -4.26 | -4.26 | -1.288 | -4.26 | -4.26 | -2.966 | -2.42 | -4.26 | -2.42 | -4.26 | 0.191 |
| 22 | -4.26 | -4.26 | -2.069 | -4.26 | -1.434 | -2.966 | -4.26 | -2.42 | -2.966 | -4.26 | -2.42 | -4.26 | 0.847 | -1.161 | 2.357 | 0.728 |