Model info
| Transcription factor | ZNF768 (GeneCards) | ||||||||
| Model | ZN768_HUMAN.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 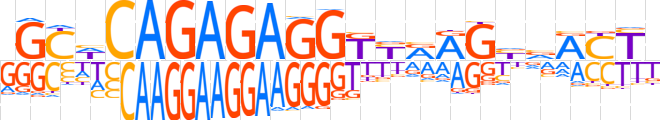 | ||||||||
| LOGO (reverse complement) | 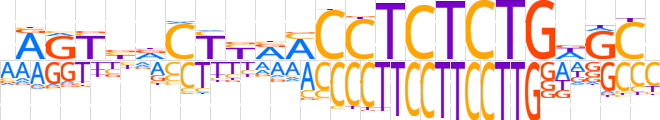 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 23 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | RGCWCAGAGAGGKYRRGbvRYTK | ||||||||
| Best auROC (human) | 0.879 | ||||||||
| Best auROC (mouse) | |||||||||
| Peak sets in benchmark (human) | 4 | ||||||||
| Peak sets in benchmark (mouse) | |||||||||
| Aligned words | 340 | ||||||||
| TF family | More than 3 adjacent zinc finger factors {2.3.3} | ||||||||
| TF subfamily | unclassified {2.3.3.0} | ||||||||
| HGNC | HGNC:26273 | ||||||||
| EntrezGene | GeneID:79724 (SSTAR profile) | ||||||||
| UniProt ID | ZN768_HUMAN | ||||||||
| UniProt AC | Q9H5H4 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | ZNF768 expression | ||||||||
| ReMap ChIP-seq dataset list | ZNF768 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 11.0 | 2.0 | 57.0 | 2.0 | 3.0 | 0.0 | 5.0 | 1.0 | 15.0 | 0.0 | 232.0 | 1.0 | 2.0 | 2.0 | 6.0 | 0.0 |
| 02 | 8.0 | 18.0 | 1.0 | 4.0 | 2.0 | 2.0 | 0.0 | 0.0 | 16.0 | 259.0 | 4.0 | 21.0 | 0.0 | 2.0 | 0.0 | 2.0 |
| 03 | 7.0 | 4.0 | 0.0 | 15.0 | 83.0 | 64.0 | 2.0 | 132.0 | 1.0 | 2.0 | 0.0 | 2.0 | 1.0 | 3.0 | 1.0 | 22.0 |
| 04 | 0.0 | 92.0 | 0.0 | 0.0 | 0.0 | 73.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 170.0 | 0.0 | 1.0 |
| 05 | 0.0 | 0.0 | 0.0 | 0.0 | 337.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 |
| 06 | 0.0 | 1.0 | 337.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 |
| 07 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 336.0 | 1.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 08 | 4.0 | 1.0 | 331.0 | 1.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 09 | 4.0 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 328.0 | 0.0 | 4.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 |
| 10 | 19.0 | 10.0 | 303.0 | 2.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 5.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 11 | 0.0 | 0.0 | 18.0 | 1.0 | 6.0 | 1.0 | 2.0 | 1.0 | 3.0 | 2.0 | 303.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 |
| 12 | 0.0 | 1.0 | 3.0 | 5.0 | 0.0 | 0.0 | 0.0 | 3.0 | 3.0 | 17.0 | 65.0 | 240.0 | 0.0 | 0.0 | 2.0 | 0.0 |
| 13 | 0.0 | 1.0 | 2.0 | 0.0 | 2.0 | 5.0 | 0.0 | 11.0 | 7.0 | 21.0 | 9.0 | 33.0 | 10.0 | 25.0 | 30.0 | 183.0 |
| 14 | 9.0 | 5.0 | 4.0 | 1.0 | 40.0 | 5.0 | 4.0 | 3.0 | 23.0 | 8.0 | 6.0 | 4.0 | 140.0 | 17.0 | 65.0 | 5.0 |
| 15 | 169.0 | 1.0 | 34.0 | 8.0 | 29.0 | 1.0 | 4.0 | 1.0 | 57.0 | 4.0 | 12.0 | 6.0 | 7.0 | 1.0 | 4.0 | 1.0 |
| 16 | 4.0 | 2.0 | 245.0 | 11.0 | 1.0 | 2.0 | 2.0 | 2.0 | 2.0 | 6.0 | 40.0 | 6.0 | 0.0 | 2.0 | 13.0 | 1.0 |
| 17 | 0.0 | 4.0 | 0.0 | 3.0 | 2.0 | 5.0 | 1.0 | 4.0 | 28.0 | 76.0 | 29.0 | 167.0 | 3.0 | 1.0 | 5.0 | 11.0 |
| 18 | 7.0 | 13.0 | 11.0 | 2.0 | 49.0 | 18.0 | 12.0 | 7.0 | 19.0 | 7.0 | 7.0 | 2.0 | 72.0 | 22.0 | 85.0 | 6.0 |
| 19 | 115.0 | 15.0 | 15.0 | 2.0 | 53.0 | 2.0 | 2.0 | 3.0 | 93.0 | 6.0 | 12.0 | 4.0 | 8.0 | 3.0 | 4.0 | 2.0 |
| 20 | 6.0 | 218.0 | 17.0 | 28.0 | 2.0 | 15.0 | 0.0 | 9.0 | 1.0 | 27.0 | 3.0 | 2.0 | 1.0 | 7.0 | 2.0 | 1.0 |
| 21 | 2.0 | 2.0 | 0.0 | 6.0 | 9.0 | 23.0 | 2.0 | 233.0 | 1.0 | 3.0 | 7.0 | 11.0 | 0.0 | 10.0 | 4.0 | 26.0 |
| 22 | 0.0 | 1.0 | 4.0 | 7.0 | 5.0 | 8.0 | 1.0 | 24.0 | 1.0 | 2.0 | 3.0 | 7.0 | 9.0 | 26.0 | 40.0 | 201.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -0.64 | -2.21 | 0.979 | -2.21 | -1.857 | -4.081 | -1.391 | -2.76 | -0.338 | -4.081 | 2.378 | -2.76 | -2.21 | -2.21 | -1.22 | -4.081 |
| 02 | -0.946 | -0.16 | -2.76 | -1.597 | -2.21 | -2.21 | -4.081 | -4.081 | -0.275 | 2.488 | -1.597 | -0.009 | -4.081 | -2.21 | -4.081 | -2.21 |
| 03 | -1.074 | -1.597 | -4.081 | -0.338 | 1.353 | 1.094 | -2.21 | 1.815 | -2.76 | -2.21 | -4.081 | -2.21 | -2.76 | -1.857 | -2.76 | 0.037 |
| 04 | -4.081 | 1.455 | -4.081 | -4.081 | -4.081 | 1.225 | -4.081 | -4.081 | -4.081 | -1.857 | -4.081 | -4.081 | -4.081 | 2.067 | -4.081 | -2.76 |
| 05 | -4.081 | -4.081 | -4.081 | -4.081 | 2.751 | -4.081 | -4.081 | -2.76 | -4.081 | -4.081 | -4.081 | -4.081 | -2.76 | -4.081 | -4.081 | -4.081 |
| 06 | -4.081 | -2.76 | 2.751 | -4.081 | -4.081 | -4.081 | -4.081 | -4.081 | -4.081 | -4.081 | -4.081 | -4.081 | -4.081 | -4.081 | -2.76 | -4.081 |
| 07 | -4.081 | -4.081 | -4.081 | -4.081 | -2.76 | -4.081 | -4.081 | -4.081 | 2.748 | -2.76 | -2.76 | -4.081 | -4.081 | -4.081 | -4.081 | -4.081 |
| 08 | -1.597 | -2.76 | 2.733 | -2.76 | -2.76 | -4.081 | -4.081 | -4.081 | -4.081 | -4.081 | -2.76 | -4.081 | -4.081 | -4.081 | -4.081 | -4.081 |
| 09 | -1.597 | -4.081 | -2.76 | -4.081 | -2.76 | -4.081 | -4.081 | -4.081 | 2.724 | -4.081 | -1.597 | -4.081 | -2.76 | -4.081 | -4.081 | -4.081 |
| 10 | -0.107 | -0.732 | 2.644 | -2.21 | -4.081 | -4.081 | -4.081 | -4.081 | -4.081 | -4.081 | -1.391 | -4.081 | -4.081 | -4.081 | -4.081 | -4.081 |
| 11 | -4.081 | -4.081 | -0.16 | -2.76 | -1.22 | -2.76 | -2.21 | -2.76 | -1.857 | -2.21 | 2.644 | -4.081 | -4.081 | -4.081 | -2.21 | -4.081 |
| 12 | -4.081 | -2.76 | -1.857 | -1.391 | -4.081 | -4.081 | -4.081 | -1.857 | -1.857 | -0.216 | 1.11 | 2.412 | -4.081 | -4.081 | -2.21 | -4.081 |
| 13 | -4.081 | -2.76 | -2.21 | -4.081 | -2.21 | -1.391 | -4.081 | -0.64 | -1.074 | -0.009 | -0.834 | 0.437 | -0.732 | 0.163 | 0.343 | 2.141 |
| 14 | -0.834 | -1.391 | -1.597 | -2.76 | 0.627 | -1.391 | -1.597 | -1.857 | 0.081 | -0.946 | -1.22 | -1.597 | 1.874 | -0.216 | 1.11 | -1.391 |
| 15 | 2.062 | -2.76 | 0.467 | -0.946 | 0.309 | -2.76 | -1.597 | -2.76 | 0.979 | -1.597 | -0.556 | -1.22 | -1.074 | -2.76 | -1.597 | -2.76 |
| 16 | -1.597 | -2.21 | 2.432 | -0.64 | -2.76 | -2.21 | -2.21 | -2.21 | -2.21 | -1.22 | 0.627 | -1.22 | -4.081 | -2.21 | -0.478 | -2.76 |
| 17 | -4.081 | -1.597 | -4.081 | -1.857 | -2.21 | -1.391 | -2.76 | -1.597 | 0.275 | 1.265 | 0.309 | 2.05 | -1.857 | -2.76 | -1.391 | -0.64 |
| 18 | -1.074 | -0.478 | -0.64 | -2.21 | 0.829 | -0.16 | -0.556 | -1.074 | -0.107 | -1.074 | -1.074 | -2.21 | 1.211 | 0.037 | 1.376 | -1.22 |
| 19 | 1.678 | -0.338 | -0.338 | -2.21 | 0.907 | -2.21 | -2.21 | -1.857 | 1.466 | -1.22 | -0.556 | -1.597 | -0.946 | -1.857 | -1.597 | -2.21 |
| 20 | -1.22 | 2.316 | -0.216 | 0.275 | -2.21 | -0.338 | -4.081 | -0.834 | -2.76 | 0.239 | -1.857 | -2.21 | -2.76 | -1.074 | -2.21 | -2.76 |
| 21 | -2.21 | -2.21 | -4.081 | -1.22 | -0.834 | 0.081 | -2.21 | 2.382 | -2.76 | -1.857 | -1.074 | -0.64 | -4.081 | -0.732 | -1.597 | 0.202 |
| 22 | -4.081 | -2.76 | -1.597 | -1.074 | -1.391 | -0.946 | -2.76 | 0.123 | -2.76 | -2.21 | -1.857 | -1.074 | -0.834 | 0.202 | 0.627 | 2.235 |