Model info
| Transcription factor | ZNF41 (GeneCards) | ||||||||
| Model | ZNF41_HUMAN.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 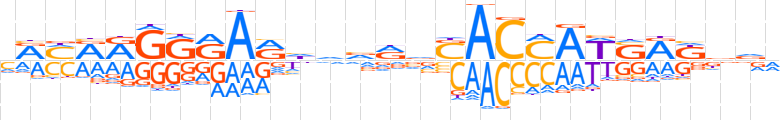 | ||||||||
| LOGO (reverse complement) | 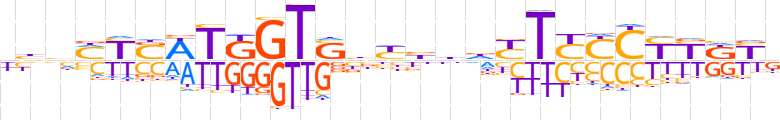 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 27 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | vRMRRGRRARhvvRvSACMAWSRKbnR | ||||||||
| Best auROC (human) | 0.919 | ||||||||
| Best auROC (mouse) | |||||||||
| Peak sets in benchmark (human) | 4 | ||||||||
| Peak sets in benchmark (mouse) | |||||||||
| Aligned words | 335 | ||||||||
| TF family | More than 3 adjacent zinc finger factors {2.3.3} | ||||||||
| TF subfamily | ZFN81-like factors {2.3.3.68} | ||||||||
| HGNC | HGNC:13107 | ||||||||
| EntrezGene | GeneID:7592 (SSTAR profile) | ||||||||
| UniProt ID | ZNF41_HUMAN | ||||||||
| UniProt AC | P51814 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | ZNF41 expression | ||||||||
| ReMap ChIP-seq dataset list | ZNF41 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 38.0 | 14.0 | 24.0 | 7.0 | 144.0 | 13.0 | 2.0 | 6.0 | 28.0 | 5.0 | 8.0 | 4.0 | 18.0 | 10.0 | 11.0 | 3.0 |
| 02 | 14.0 | 192.0 | 7.0 | 15.0 | 20.0 | 4.0 | 7.0 | 11.0 | 6.0 | 26.0 | 8.0 | 5.0 | 1.0 | 6.0 | 6.0 | 7.0 |
| 03 | 12.0 | 14.0 | 11.0 | 4.0 | 198.0 | 6.0 | 10.0 | 14.0 | 16.0 | 1.0 | 9.0 | 2.0 | 10.0 | 5.0 | 19.0 | 4.0 |
| 04 | 170.0 | 3.0 | 58.0 | 5.0 | 16.0 | 2.0 | 0.0 | 8.0 | 24.0 | 1.0 | 23.0 | 1.0 | 9.0 | 0.0 | 14.0 | 1.0 |
| 05 | 15.0 | 0.0 | 195.0 | 9.0 | 1.0 | 0.0 | 4.0 | 1.0 | 16.0 | 3.0 | 70.0 | 6.0 | 2.0 | 2.0 | 11.0 | 0.0 |
| 06 | 11.0 | 6.0 | 15.0 | 2.0 | 5.0 | 0.0 | 0.0 | 0.0 | 21.0 | 10.0 | 230.0 | 19.0 | 4.0 | 1.0 | 11.0 | 0.0 |
| 07 | 13.0 | 0.0 | 27.0 | 1.0 | 9.0 | 1.0 | 5.0 | 2.0 | 104.0 | 5.0 | 146.0 | 1.0 | 5.0 | 0.0 | 16.0 | 0.0 |
| 08 | 123.0 | 2.0 | 5.0 | 1.0 | 4.0 | 0.0 | 0.0 | 2.0 | 188.0 | 0.0 | 3.0 | 3.0 | 2.0 | 0.0 | 1.0 | 1.0 |
| 09 | 141.0 | 16.0 | 157.0 | 3.0 | 2.0 | 0.0 | 0.0 | 0.0 | 6.0 | 0.0 | 3.0 | 0.0 | 1.0 | 2.0 | 4.0 | 0.0 |
| 10 | 46.0 | 44.0 | 12.0 | 48.0 | 12.0 | 2.0 | 0.0 | 4.0 | 27.0 | 23.0 | 14.0 | 100.0 | 2.0 | 0.0 | 1.0 | 0.0 |
| 11 | 29.0 | 21.0 | 27.0 | 10.0 | 45.0 | 7.0 | 5.0 | 12.0 | 9.0 | 8.0 | 6.0 | 4.0 | 70.0 | 23.0 | 29.0 | 30.0 |
| 12 | 84.0 | 22.0 | 34.0 | 13.0 | 43.0 | 4.0 | 6.0 | 6.0 | 23.0 | 20.0 | 16.0 | 8.0 | 13.0 | 8.0 | 31.0 | 4.0 |
| 13 | 51.0 | 9.0 | 89.0 | 14.0 | 38.0 | 1.0 | 9.0 | 6.0 | 33.0 | 3.0 | 46.0 | 5.0 | 9.0 | 1.0 | 17.0 | 4.0 |
| 14 | 37.0 | 17.0 | 67.0 | 10.0 | 7.0 | 3.0 | 2.0 | 2.0 | 45.0 | 51.0 | 49.0 | 16.0 | 4.0 | 6.0 | 11.0 | 8.0 |
| 15 | 2.0 | 65.0 | 15.0 | 11.0 | 6.0 | 62.0 | 3.0 | 6.0 | 4.0 | 90.0 | 28.0 | 7.0 | 3.0 | 24.0 | 3.0 | 6.0 |
| 16 | 15.0 | 0.0 | 0.0 | 0.0 | 240.0 | 0.0 | 1.0 | 0.0 | 47.0 | 0.0 | 2.0 | 0.0 | 25.0 | 1.0 | 4.0 | 0.0 |
| 17 | 7.0 | 300.0 | 16.0 | 4.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 2.0 | 4.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 18 | 3.0 | 1.0 | 2.0 | 1.0 | 59.0 | 231.0 | 0.0 | 13.0 | 13.0 | 4.0 | 2.0 | 1.0 | 4.0 | 0.0 | 0.0 | 1.0 |
| 19 | 47.0 | 2.0 | 27.0 | 3.0 | 227.0 | 5.0 | 3.0 | 1.0 | 1.0 | 0.0 | 2.0 | 1.0 | 7.0 | 0.0 | 8.0 | 1.0 |
| 20 | 27.0 | 8.0 | 20.0 | 227.0 | 4.0 | 1.0 | 1.0 | 1.0 | 9.0 | 5.0 | 8.0 | 18.0 | 2.0 | 0.0 | 1.0 | 3.0 |
| 21 | 5.0 | 6.0 | 29.0 | 2.0 | 6.0 | 3.0 | 1.0 | 4.0 | 5.0 | 5.0 | 17.0 | 3.0 | 20.0 | 32.0 | 185.0 | 12.0 |
| 22 | 25.0 | 0.0 | 11.0 | 0.0 | 38.0 | 1.0 | 2.0 | 5.0 | 176.0 | 12.0 | 17.0 | 27.0 | 10.0 | 0.0 | 9.0 | 2.0 |
| 23 | 21.0 | 16.0 | 183.0 | 29.0 | 5.0 | 4.0 | 4.0 | 0.0 | 7.0 | 7.0 | 18.0 | 7.0 | 2.0 | 5.0 | 22.0 | 5.0 |
| 24 | 8.0 | 1.0 | 19.0 | 7.0 | 6.0 | 6.0 | 2.0 | 18.0 | 36.0 | 40.0 | 104.0 | 47.0 | 0.0 | 19.0 | 11.0 | 11.0 |
| 25 | 11.0 | 7.0 | 28.0 | 4.0 | 39.0 | 6.0 | 9.0 | 12.0 | 40.0 | 15.0 | 70.0 | 11.0 | 12.0 | 14.0 | 42.0 | 15.0 |
| 26 | 85.0 | 4.0 | 10.0 | 3.0 | 17.0 | 13.0 | 4.0 | 8.0 | 109.0 | 15.0 | 13.0 | 12.0 | 11.0 | 4.0 | 22.0 | 5.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 0.588 | -0.394 | 0.134 | -1.062 | 1.914 | -0.466 | -2.199 | -1.208 | 0.286 | -1.379 | -0.935 | -1.586 | -0.148 | -0.72 | -0.628 | -1.846 |
| 02 | -0.394 | 2.201 | -1.062 | -0.327 | -0.045 | -1.586 | -1.062 | -0.628 | -1.208 | 0.213 | -0.935 | -1.379 | -2.749 | -1.208 | -1.208 | -1.062 |
| 03 | -0.544 | -0.394 | -0.628 | -1.586 | 2.231 | -1.208 | -0.72 | -0.394 | -0.264 | -2.749 | -0.822 | -2.199 | -0.72 | -1.379 | -0.095 | -1.586 |
| 04 | 2.079 | -1.846 | 1.008 | -1.379 | -0.264 | -2.199 | -4.071 | -0.935 | 0.134 | -2.749 | 0.092 | -2.749 | -0.822 | -4.071 | -0.394 | -2.749 |
| 05 | -0.327 | -4.071 | 2.216 | -0.822 | -2.749 | -4.071 | -1.586 | -2.749 | -0.264 | -1.846 | 1.195 | -1.208 | -2.199 | -2.199 | -0.628 | -4.071 |
| 06 | -0.628 | -1.208 | -0.327 | -2.199 | -1.379 | -4.071 | -4.071 | -4.071 | 0.003 | -0.72 | 2.381 | -0.095 | -1.586 | -2.749 | -0.628 | -4.071 |
| 07 | -0.466 | -4.071 | 0.25 | -2.749 | -0.822 | -2.749 | -1.379 | -2.199 | 1.589 | -1.379 | 1.927 | -2.749 | -1.379 | -4.071 | -0.264 | -4.071 |
| 08 | 1.756 | -2.199 | -1.379 | -2.749 | -1.586 | -4.071 | -4.071 | -2.199 | 2.18 | -4.071 | -1.846 | -1.846 | -2.199 | -4.071 | -2.749 | -2.749 |
| 09 | 1.893 | -0.264 | 2.0 | -1.846 | -2.199 | -4.071 | -4.071 | -4.071 | -1.208 | -4.071 | -1.846 | -4.071 | -2.749 | -2.199 | -1.586 | -4.071 |
| 10 | 0.778 | 0.734 | -0.544 | 0.82 | -0.544 | -2.199 | -4.071 | -1.586 | 0.25 | 0.092 | -0.394 | 1.55 | -2.199 | -4.071 | -2.749 | -4.071 |
| 11 | 0.321 | 0.003 | 0.25 | -0.72 | 0.756 | -1.062 | -1.379 | -0.544 | -0.822 | -0.935 | -1.208 | -1.586 | 1.195 | 0.092 | 0.321 | 0.354 |
| 12 | 1.376 | 0.049 | 0.478 | -0.466 | 0.711 | -1.586 | -1.208 | -1.208 | 0.092 | -0.045 | -0.264 | -0.935 | -0.466 | -0.935 | 0.387 | -1.586 |
| 13 | 0.88 | -0.822 | 1.434 | -0.394 | 0.588 | -2.749 | -0.822 | -1.208 | 0.449 | -1.846 | 0.778 | -1.379 | -0.822 | -2.749 | -0.204 | -1.586 |
| 14 | 0.562 | -0.204 | 1.151 | -0.72 | -1.062 | -1.846 | -2.199 | -2.199 | 0.756 | 0.88 | 0.84 | -0.264 | -1.586 | -1.208 | -0.628 | -0.935 |
| 15 | -2.199 | 1.121 | -0.327 | -0.628 | -1.208 | 1.074 | -1.846 | -1.208 | -1.586 | 1.445 | 0.286 | -1.062 | -1.846 | 0.134 | -1.846 | -1.208 |
| 16 | -0.327 | -4.071 | -4.071 | -4.071 | 2.423 | -4.071 | -2.749 | -4.071 | 0.799 | -4.071 | -2.199 | -4.071 | 0.175 | -2.749 | -1.586 | -4.071 |
| 17 | -1.062 | 2.646 | -0.264 | -1.586 | -4.071 | -2.749 | -4.071 | -4.071 | -4.071 | -2.199 | -1.586 | -2.749 | -4.071 | -4.071 | -4.071 | -4.071 |
| 18 | -1.846 | -2.749 | -2.199 | -2.749 | 1.025 | 2.385 | -4.071 | -0.466 | -0.466 | -1.586 | -2.199 | -2.749 | -1.586 | -4.071 | -4.071 | -2.749 |
| 19 | 0.799 | -2.199 | 0.25 | -1.846 | 2.368 | -1.379 | -1.846 | -2.749 | -2.749 | -4.071 | -2.199 | -2.749 | -1.062 | -4.071 | -0.935 | -2.749 |
| 20 | 0.25 | -0.935 | -0.045 | 2.368 | -1.586 | -2.749 | -2.749 | -2.749 | -0.822 | -1.379 | -0.935 | -0.148 | -2.199 | -4.071 | -2.749 | -1.846 |
| 21 | -1.379 | -1.208 | 0.321 | -2.199 | -1.208 | -1.846 | -2.749 | -1.586 | -1.379 | -1.379 | -0.204 | -1.846 | -0.045 | 0.418 | 2.164 | -0.544 |
| 22 | 0.175 | -4.071 | -0.628 | -4.071 | 0.588 | -2.749 | -2.199 | -1.379 | 2.114 | -0.544 | -0.204 | 0.25 | -0.72 | -4.071 | -0.822 | -2.199 |
| 23 | 0.003 | -0.264 | 2.153 | 0.321 | -1.379 | -1.586 | -1.586 | -4.071 | -1.062 | -1.062 | -0.148 | -1.062 | -2.199 | -1.379 | 0.049 | -1.379 |
| 24 | -0.935 | -2.749 | -0.095 | -1.062 | -1.208 | -1.208 | -2.199 | -0.148 | 0.535 | 0.639 | 1.589 | 0.799 | -4.071 | -0.095 | -0.628 | -0.628 |
| 25 | -0.628 | -1.062 | 0.286 | -1.586 | 0.614 | -1.208 | -0.822 | -0.544 | 0.639 | -0.327 | 1.195 | -0.628 | -0.544 | -0.394 | 0.688 | -0.327 |
| 26 | 1.388 | -1.586 | -0.72 | -1.846 | -0.204 | -0.466 | -1.586 | -0.935 | 1.636 | -0.327 | -0.466 | -0.544 | -0.628 | -1.586 | 0.049 | -1.379 |